Estudios recientes de asociación de genoma completo han identificado un locus en el cromosoma 12q.13.3 asociado con las concentraciones plasmáticas de triglicéridos y colesterol unido a lipoproteínas de alta densidad; rs11613352 es el polimorfismo de un solo nucleótido líder en dicho locus. El objetivo del estudio es investigar la implicación de rs11613352 en una población con elevado riesgo cardiovascular por hipercolesterolemia familiar.
MétodosSe genotipificó mediante análisis Taqman® el polimorfismo de un solo nucleótido en una cohorte de 601 pacientes con hipercolesterolemia familiar no relacionados, y se analizó su asociación genética con las concentraciones plasmáticas de triglicéridos y colesterol unido a lipoproteínas de alta densidad, mediante métodos multivariables basados en regresión lineal.
ResultadosLa frecuencia alélica mínima fue de 0,17 y las frecuencias genotípicas, 0,69, 0,27 y 0,04 para los genotipos CC, CT y TT respectivamente. El polimorfismo se asoció de manera recesiva (genotipo TT) con disminución de los triglicéridos (p=0,002) y aumento de colesterol unido a lipoproteínas de alta densidad (p=0,021) tras ajustar por edad y sexo.
ConclusionesEl polimorfismo rs11613352 puede contribuir a modular el riesgo cardiovascular al modificar las concentraciones plasmáticas de lípidos en los pacientes con hipercolesterolemia familiar.
Palabras clave
La hipercolesterolemia familiar (HF) es uno de los trastornos hereditarios más frecuentes, y se caracteriza por una elevación intensa del colesterol unido a las lipoproteínas de baja densidad (LDL) en plasma y una enfermedad aterosclerótica prematura. Las mutaciones del gen del receptor de LDL clásico son la principal causa de esta enfermedad. Sin embargo, hay diferencias sustanciales en el inicio y la gravedad de la aterosclerosis en los pacientes con HF heterocigotos, lo cual podría deberse a factores ambientales y metabólicos y otros factores genéticos1,2.
De entre los genes y regiones candidatos involucrados en el metabolismo lipídico, el polimorfismo en el locus del cromosoma rs11613352 se halló en estudios de asociación de genoma completo (GWAS) como polimorfismo líder en el locus del cromosoma 12q13.3 asociado a una disminución significativa de los triglicéridos (TG) y un aumento de las lipoproteínas de alta densidad (HDL) en plasma3, así como a unos valores séricos de ácido úrico inferiores4.
El objetivo del estudio es analizar por primera vez la asociación de este polimorfismo con las concentraciones de lípidos en plasma en esta población propensa a la aterosclerosis (HF).
MÉTODOSPoblación estudiadaSe incluyó en el estudio a 601 pacientes no emparentados, casos índice de familias con HF seleccionados aleatoriamente del Spanish FH Longitudinal Cohort Study (SAFEHEART). Las principales características de la cohorte de HF se han descrito ya5,6. Se obtuvieron de cada participante datos antropométricos como el índice de masa corporal, perímetro de cintura, cociente cintura/cadera y la presencia de arco corneal, xantomas, diabetes mellitus, hipertensión y hábito tabáquico, junto con las determinaciones bioquímicas de lípidos en plasma de cada sujeto obtenidas según métodos previamente descritos5,6 y que se presentan con mayor detalle en el .
Se consideró positivos para las manifestaciones clínicas de enfermedad cardiovascular (ECV) a los pacientes con HF si tenían antecedentes documentados de infarto de miocardio, cirugía de bypass arterial coronario, angioplastia coronaria transluminal percutánea, angina de pecho con aterosclerosis coronaria angiográfica (estenosis > 50%), ictus aterotrombótico isquémico o enfermedad vascular arterial periférica crónica.
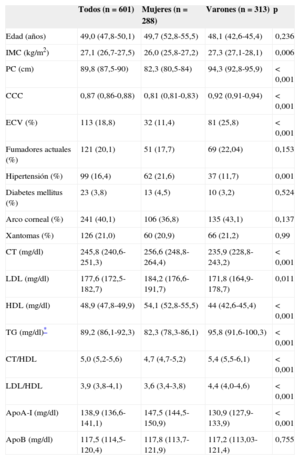
Todos los pacientes incluidos en el estudio eran portadores heterocigotos de mutaciones en el receptor de LDL (gen LDLR)7. Las características bioquímicas y clínicas de la población con HF se presentan en la tabla 1.
Características iniciales de la población con hipercolesterolemia familiar estudiada
| Todos (n=601) | Mujeres (n=288) | Varones (n=313) | p | |
|---|---|---|---|---|
| Edad (años) | 49,0 (47,8-50,1) | 49,7 (52,8-55,5) | 48,1 (42,6-45,4) | 0,236 |
| IMC (kg/m2) | 27,1 (26,7-27,5) | 26,0 (25,8-27,2) | 27,3 (27,1-28,1) | 0,006 |
| PC (cm) | 89,8 (87,5-90) | 82,3 (80,5-84) | 94,3 (92,8-95,9) | <0,001 |
| CCC | 0,87 (0,86-0,88) | 0,81 (0,81-0,83) | 0,92 (0,91-0,94) | < 0,001 |
| ECV (%) | 113 (18,8) | 32 (11,4) | 81 (25,8) | < 0,001 |
| Fumadores actuales (%) | 121 (20,1) | 51 (17,7) | 69 (22,04) | 0,153 |
| Hipertensión (%) | 99 (16,4) | 62 (21,6) | 37 (11,7) | 0,001 |
| Diabetes mellitus (%) | 23 (3,8) | 13 (4,5) | 10 (3,2) | 0,524 |
| Arco corneal (%) | 241 (40,1) | 106 (36,8) | 135 (43,1) | 0,137 |
| Xantomas (%) | 126 (21,0) | 60 (20,9) | 66 (21,2) | 0,99 |
| CT (mg/dl) | 245,8 (240,6-251,3) | 256,6 (248,8-264,4) | 235,9 (228,8-243,2) | < 0,001 |
| LDL (mg/dl) | 177,6 (172,5-182,7) | 184,2 (176,6-191,7) | 171,8 (164,9-178,7) | 0,011 |
| HDL (mg/dl) | 48,9 (47,8-49,9) | 54,1 (52,8-55,5) | 44 (42,6-45,4) | < 0,001 |
| TG (mg/dl)* | 89,2 (86,1-92,3) | 82,3 (78,3-86,1) | 95,8 (91,6-100,3) | < 0,001 |
| CT/HDL | 5,0 (5,2-5,6) | 4,7 (4,7-5,2) | 5,4 (5,5-6,1) | < 0,001 |
| LDL/HDL | 3,9 (3,8-4,1) | 3,6 (3,4-3,8) | 4,4 (4,0-4,6) | < 0,001 |
| ApoA-I (mg/dl) | 138,9 (136,6-141,1) | 147,5 (144,5-150,9) | 130,9 (127,9-133,9) | < 0,001 |
| ApoB (mg/dl) | 117,5 (114,5-120,4) | 117,8 (113,7-121,9) | 117,2 (113,03-121,4) | 0,755 |
ApoA-I: apolipoproteína A-I; ApoB: apolipoproteína B; CCC: cociente cintura/cadera; CT: colesterol total; ECV: enfermedad cardiovascular; HDL: lipoproteínas de alta densidad; IM: índice de masa corporal; LDL: lipoproteínas de baja densidad; PC: perímetro de cintura; TG: triglicéridos.
Los datos expresan media (intervalo de confianza del 95%) o n. (%).
La significación estadística se probó mediante la prueba de la t de Student o la prueba exacta de Fisher, respectivamente.
La significación estadística se estableció en un valor de p < 0,05.
Se obtuvo ADN a partir de muestras de sangre total empleando el kit Wizard DNA Purification de Promega. Se determinó el gentipo del polimorfismo rs11613352 mediante un análisis Taqman® específico para el alelo utilizado en el Applied Biosystems® 7900 Real-Time PCR System. La discriminación alélica se realizó con un ABI PRISM® 7900, al igual que para la determinación del genotipo.
Análisis estadísticoLos datos cuantitativos se expresan en forma de media ± des-viación estándar y los datos categóricos, en porcentajes. Se aplicó una transformación logarítmica a los valores de TG para el análisis estadístico. La significación estadística se evaluó con la prueba de la t de Student para datos cuantitativos y con la prueba exacta de Fisher o la prueba de la χ2 para las variables discretas.
Se calcularon las frecuencias alélicas a partir de los genotipos de los participantes. La distribución genotípica en la población se evaluó respecto al equilibrio de Hardy-Weinberg mediante la prueba de la χ2. Las diferencias cuantitativas de los valores de lípidos entre los genotipos se ajustaron respecto a edad y sexo, se evaluaron mediante regresión lineal y se resumieron con media, error estándar, diferencia media respecto a una categoría de referencia e intervalo de confianza del 95% (IC95%) de las diferencias. Se evaluaron diversos modelos de transmisión hereditaria (modelo general o codominante, dominante y recesivo), y se calcularon los efectos genéticos para cada genotipo en comparación con el genotipo de referencia de cada modelo. El análisis estadístico se realizó con el programa SPSS 19.0 (SPSS Inc.; Chicago, Illinois, Estados Unidos) y el programa SNPStats8. Se consideraron significativos los valores de p<0,05. La potencia estadística para detectar diferencias significativas entre los grupos de genotipos en cuanto a concentración de lípidos en plasma se analizó con el programa Power Calculator (Quantor v2.1).
RESULTADOSCaracterísticas clínicas y bioquímicas de la población con hipercolesterolemia familiar estudiadaLas características basales de la población con HF estudiada (el 48% mujeres y el 52% varones) se presentan en la tabla 1. La mayoría de los pacientes (87%) recibían tratamiento hipolipemiante en el momento de la inclusión en el estudio y no hubo diferencias de tratamiento entre los distintos grupos de genotipos (datos no presentados).
Asociación del rs11613352 con las concentraciones de triglicéridos y lipoproteínas de alta densidadLas frecuencias genotípicas observadas son compatibles con el equilibrio de Hardy-Weinberg (p=0,32). La frecuencia de los alelos C y T en la HF fue 0,83 y 0,17 respectivamente. Las frecuencias de los genotipos CC, CT y TT fueron del 69, el 27 y el 4% respectivamente.
No hubo diferencias en las frecuencias de genotipos en función del sexo o rasgos antropométricos como el perímetro de cintura, el cociente cintura/cadera o el índice de masa corporal, ni en relación con los antecedentes de ECV, el hábito tabáquico actual o los valores de lípidos distintos del TG y las HDL (prueba de la t de Student, p=0,003 y p=0,007 respectivamente). Sin embargo, es interesante señalar que no se observó ningún genotipo TT en los pacientes con xantomas (prueba exacta de Fisher, p=0,012) ().
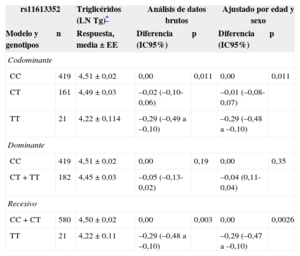
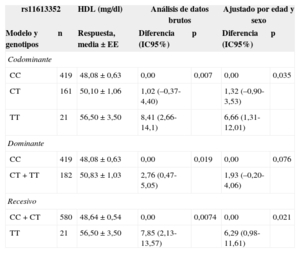
La asociación genotípica con las concentraciones de lípidos en plasma solo fue significativa las de TG y HDL según lo ya descrito3 (tablas 2 y 3 respectivamente). La mejor asociación fue la alcanzada con un modelo de transmisión hereditaria recesiva para los valores plasmáticos de TG y HDL. El análisis de los datos brutos de la asociación genética mostró asociación significativa del genotipo TT con disminución de TG (p=0,003) y aumento de HDL en plasma (p=0,0074). La asociación continúa siendo significativa tras introducir un ajuste para edad y sexo: p=0,0026; diferencia media, –0,29 (IC95%, –0,48 a –0,10) para el logaritmo natural de los valores de TG, que corresponde a un cambio de 22mg/dl entre los genotipos (tabla 2); y p=0,021; diferencia media, 6,29 (IC95%, 0,98-11,61) mg/dl para las HDL (tabla 3).
Asociación de rs11613352 con las concentraciones de triglicéridos
| rs11613352 | Triglicéridos (LN Tg)* | Análisis de datos brutos | Ajustado por edad y sexo | |||
|---|---|---|---|---|---|---|
| Modelo y genotipos | n | Respuesta, media±EE | Diferencia (IC95%) | p | Diferencia (IC95%) | p |
| Codominante | ||||||
| CC | 419 | 4,51±0,02 | 0,00 | 0,011 | 0,00 | 0,011 |
| CT | 161 | 4,49±0,03 | –0,02 (–0,10-0,06) | –0,01 (–0,08-0,07) | ||
| TT | 21 | 4,22±0,114 | –0,29 (–0,49 a –0,10) | –0,29 (–0,48 a –0,10) | ||
| Dominante | ||||||
| CC | 419 | 4,51±0,02 | 0,00 | 0,19 | 0,00 | 0,35 |
| CT+TT | 182 | 4,45±0,03 | –0,05 (–0,13-0,02) | –0,04 (0,11-0,04) | ||
| Recesivo | ||||||
| CC+CT | 580 | 4,50±0,02 | 0,00 | 0,003 | 0,00 | 0,0026 |
| TT | 21 | 4,22±0,11 | –0,29 (–0,48 a –0,10) | –0,29 (–0,47 a –0,10) | ||
EE: error estándar; IC95%: intervalo de confianza del 95%.
Asociación de rs11613352 con las concentraciones de lipoproteínas de alta densidad
| rs11613352 | HDL (mg/dl) | Análisis de datos brutos | Ajustado por edad y sexo | |||
|---|---|---|---|---|---|---|
| Modelo y genotipos | n | Respuesta, media±EE | Diferencia (IC95%) | p | Diferencia (IC95%) | p |
| Codominante | ||||||
| CC | 419 | 48,08±0,63 | 0,00 | 0,007 | 0,00 | 0,035 |
| CT | 161 | 50,10±1,06 | 1,02 (–0,37-4,40) | 1,32 (–0,90-3,53) | ||
| TT | 21 | 56,50±3,50 | 8,41 (2,66-14,1) | 6,66 (1,31-12,01) | ||
| Dominante | ||||||
| CC | 419 | 48,08±0,63 | 0,00 | 0,019 | 0,00 | 0,076 |
| CT+TT | 182 | 50,83±1,03 | 2,76 (0,47-5,05) | 1,93 (–0,20-4,06) | ||
| Recesivo | ||||||
| CC+CT | 580 | 48,64±0,54 | 0,00 | 0,0074 | 0,00 | 0,021 |
| TT | 21 | 56,50±3,50 | 7,85 (2,13-13,57) | 6,29 (0,98-11,61) | ||
EE: error estándar; HDL: lipoproteínas de alta densidad; IC95%: intervalo de confianza del 95%.
En los cálculos de la potencia estadística se asumió la independencia de los individuos, una mínima frecuencia del alelo de 0,17, un efecto genético recesivo, una media poblacional de 4,49 y una desviación estándar de 0,48 para el logaritmo natural de TG, y un valor medio de HDL de 48,9mg/dl con una desviación estándar de 13,2mg/dl y un error de tipo I de 0,05 (unilateral). Con 601 individuos con HF, había una potencia estadística del 80% para detectar una magnitud del efecto de –0,3 para los TG, lo cual corresponde a un cambio de aproximadamente 22 mg/dl respecto a la media de la población, y a una potencia de más del 70% para detectar una magnitud del efecto > 7 mg/dl respecto al valor medio de HDL.
DISCUSIÓNEstudiar los nuevos genes candidatos hallados por GWAS y sus polimorfismos podría ser útil para comprender la variabilidad fenotípica en la aparición de las manifestaciones clínicas de la ECV en los pacientes con HF. Además, puede tener interés para un futuro uso terapéutico contra la aterosclerosis.
Las LDL elevadas, los TG elevados y las HDL reducidas son importantes factores de riesgo de enfermedad coronaria9–11. No solo las concentraciones de lipoproteínas intervienen en la aterogénesis, sino también el tamaño y la calidad de las partículas lipoproteicas. El tamaño de las partículas de LDL y HDL está relacionado con la concentración plasmática de TG y el cociente TG/HDL muestra buena correlación con el tamaño de las partículas de HDL y con la enfermedad coronaria7. En la HF, los pacientes con una elevación de los TG (> 150mg/dl) presentan más partículas de HDL aterógenas, con una capacidad antiinflamatoria reducida y una disminución de la capacidad de fomentar la salida de colesterol de los macrófagos respecto a individuos normales12. Así pues, las variantes genéticas que afectan a las HDL y los TG pueden ser de gran relevancia en el proceso aterosclerótico. Sin embargo, recientemente los estudios de aleatorización genética han puesto en duda el papel de las mutaciones que afectan básicamente a los valores de HDL en la ECV13.
En los GWAS, se han observado variantes genéticas frecuentes y variantes raras en genes candidatos que contribuyen a la dislipemia, si bien continúa sin conocerse gran parte de la variabilidad14,15. El polimorfismo de GWAS rs11613352, recientemente descubierto, se asoció principalmente con los valores de TG3 y también con los de HDL. Este estudio, llevado a cabo en una población con HF, pone de manifiesto por primera vez que el polimorfismo rs11613352 se asocia a unas cifras de TG y HDL variables. El genotipo TT se asocia a un perfil beneficioso de valores de TG inferiores y de HDL más altos.
El posible efecto protector del genotipo TT se pone de relieve también en la ausencia total de xantomas tendinosos en los pacientes con HF que tienen este genotipo; la presencia de ECV tuvo una prevalencia de casi la mitad entre los pacientes con el genotipo TT (), aunque el tamaño de la muestra no produjo significación estadística.
Hay varios genes que se encuentran en la región de ±500 kb de la señal de GWAS del rs11613352 que indicadores de TG3. Entre los genes candidatos de interés en esta región (), se encuentran el de la proteína relacionada con el receptor de LDL 1 (LRP1)3, que forma parte de la familia de receptores de LDL3,6,16; los genes de la región intergénica R3HDM2-INHBC-INHBE (12q1,31-33)4, que codifican la proteína mal caracterizada R3HDM2 (KIAA1002), un dominio R3H que se ha demostrado que se une al ácido nucleico monocatenario17, y los genes INHBC e INHBE de la superfamilia TGFβ, que incluyen entre sus múltiples funciones un papel en la regulación del metabolismo, la homeostasis y las respuestas inmunitarias; recientemente se ha identificado el ARHGAP9 como gen candidato en el tejido adiposo18. Por el momento, no existen datos funcionales que relacionen alguno de los genes situados en la proximidad de rs11613352 con las concentraciones lipídicas.
El LRP1 es un receptor que interviene en la captación de los lípidos17,19–21 y está situado a unas 200kb del rs11613352. En un estudio previo de asociación genética con variantes de LRP1 en población con HF6, no se observó asociación de 10 polimorfismos de un solo nucleótido del LRP1 con los valores de lípidos plasmáticos en ayunas (colesterol total, LDL, HDL y TG) en la HF (datos no presentados); tampoco se ha observado un desequilibrio de ligamiento entre estos polimorfismos de un solo nucleótido del LRP1 y el polimorfismo líder de GWAS para TG, rs11613352; por consiguiente, no se cree que el LRP1 sea el gen candidato involucrado en la modulación de las concentraciones plasmáticas de TG y HDL.
CONCLUSIONESSe ha observado que el polimorfismo rs11613352 puede contribuir a atenuar el riesgo cardiovascular modificando las concentraciones plasmáticas de lípidos de los pacientes con HF, puesto que el genotipo TT presenta un perfil de menos TG y más HDL. Los efectos concuerdan con los resultados previos de estudios a gran escala de GWAS sobre las concentraciones de lípidos3,4, aunque sería necesaria una confirmación que reproduzca los resultados en otras poblaciones HF independientes.
El rs11613352 está situado dentro del intrón 1 del gen mal conocido R3HDM2 (o KIAAA1002) y cerca del cluster transcripcionalmente opuesto INHBC-INHBE, una región rica en centros reguladores transcripcionales. Serán necesarios nuevos estudios de expresión para esclarecer el (los) gen(es) afectados por esta variante genética y su función molecular en la modulación de las conentraciones plasmáticas de lípidos y el riesgo de ECV.
FINANCIACIÓNEste estudio fue financiado en parte por SAF (Servicio de Actividad Física) —SAF2010-16549—, CNIC (Centro Nacional de Investigaciones Cardiovasculares) —CNIC-08-2008—, RIC (Red de Investigación Cardiovascular) RD12/0042/0027 y TerCel (Red de Terapia Celular) —RD12/0019/0026— del ISCIII (Instituto de Salud Carlos III), España.
CONFLICTO DE INTERESESNinguno.
Damos las gracias a la Fundación Hipercolesterolemia Familiar de España por su ayuda en el reclutamiento de participantes y controles, así como a los pacientes con HF por su valiosa contribución y su disposición a participar en el estudio. Agradecemos a Montse Gómez-Pardo su ayuda técnica y a la Fundación de Investigación Cardiovascular-Fundación Jesús Serra su apoyo para el estudio.