La expresión del gen LRP1, muy acentuada en la placa aterosclerótica, se asocia con un aumento en la incorporación de lípidos por la pared vascular. Nuestro objetivo es analizar si el gen LRP1 modula el riesgo genético de aparición de enfermedad cardiovascular prematura en pacientes con hipercolesterolemia familiar mediante asociación de polimorfismos.
MétodosSe genotipificaron 10 polimorfismos de un solo nucleótido del gen LRP1 (rs715948, rs1799986, rs1800127, rs7968719, rs1800176, rs1800194, rs1800181, rs1140648, rs1800164 y rs35282763) en 339 pacientes (77 con enfermedad cardiovascular y 262 sin enfermedad) pertenecientes al estudio SAFEHEART.
ResultadosSe halló una asociacion significativa con el polimorfismo c.677C>T (rs1799986) tras ajustar por sexo, edad, índice de masa corporal y efecto de la mutación del receptor de lipoproteínas de baja densidad con el modelo dominante (CT+TT frente a CC: odds ratio=1,94; intervalo de confianza del 95%, 1,08-3,48; p=0,029). Tras ampliar la población a 648 individuos (133 con enfermedad y 515 sin ella), se obtuvieron resultados similares (odds ratio=1,83; intervalo de confianza del 95%, 1,16-2,88; p=0,011).
ConclusionesEl polimorfismo c.677C>T se asocia con aumento del riesgo de enfermedad cardiovascular prematura en la hipercolesterolemia familiar. Aunque su implicación en la alteración de un patrón normal de procesamiento del ARNm no se ha corroborado, no se descarta que dicho polimorfismo se halle en desequilibrio de ligamento con otro polimorfismo funcional en el que resida la relación causa-efecto con la enfermedad cardiovascular. Serían necesarios más estudios para corroborar los resultados y localizar las variantes genéticas relacionadas con dicho polimorfismo implicadas en conferir riesgo de enfermedad cardiovascular.
Palabras clave
La hipercolesterolemia familiar (HF) se caracteriza por alta concentración plasmática de colesterol unido a lipoproteínas de baja densidad, lo que causa un exceso de depósitos de colesterol en los tejidos y conduce a una aterosclerosis acelerada y un aumento del riesgo de padecer una enfermedad cardiovascular prematura (ECVp)1. La anomalía genética más frecuente es las mutaciones en el receptor de lipoproteínas de baja densidad (LDLR); la frecuencia poblacional de hipercolesterolemia familiar heterocigótica (HFh) es de aproximadamente 1/500. Aunque la causa genética de la enfermedad son las mutaciones en el gen LDLR, el fenotipo clínico de la HF es variable independientemente del tipo de mutación, por lo que se asume que dicha variabilidad se debe a factores tanto ambientales como genéticos2, 3.
El descubrimiento de variaciones en otros genes que puedan modificar la predisposición a sufrir un evento cardiovascular es de gran interés por su posible utilización como marcadores predictores de la enfermedad4. Los genes implicados en la incorporación y la homeostasis de colesterol son buenos candidatos de estudio. La proteína 1 relacionada con el receptor de lipoproteínas de baja densidad (LRP1) es un receptor transmembrana multiligando de la familia de LDLR que se une y facilita la eliminación de una gran variedad de ligandos implicados en la fibrinolisis, la aterogénesis y la trombogénesis5, 6, 7. La sobreexpresión del LRP1 en la placa aterosclerótica se ha demostrado tanto en modelos animales como en humanos8, 9. Diversos estudios han señalado relación entre alteraciones en la expresión del LRP1 y enfermedad coronaria8, 10. Asimismo se ha descrito una expresión acentuada en la proteína como en el ARNm de pacientes con HF homocigotos11. En conjunto, los resultados publicados indican que el LRP1 puede ser un receptor clave en la etiopatogenia de la aterosclerosis.
El objetivo de este estudio es analizar si hay polimorfismos del gen LRP1 que se asocian con la aparición de ECVp en la HF.
MétodosPoblaciónEl estudio incluyó inicialmente 339 casos no relacionados de pacientes heterocigotos con HF procedentes del estudio SAFEHEART12, 13 («Métodos» del material adicional). Se diagnosticó enfermedad cardiovascular (ECV) si el paciente tenía antecedente de infarto de miocardio, bypass coronario o angioplastia, angina de pecho con aterosclerosis coronaria diagnosticada angiográficamente (estenosis>50%), ictus o evento vascular periférico. Se consideró ECVp si el evento ocurrió antes de los 55 años de edad en varones y antes de los 65 en mujeres14. De cada paciente, se recogieron datos sobre concentraciones lipídicas en sangre, hipertensión arterial, diabetes y tabaquismo y datos antropométricos. Las características de la población estudiada se muestran en la Tabla 1. Todos los pacientes incluidos tenían identificadas mutaciones en el LDLR. Las mutaciones halladas en los individuos analizados y su clasificación según actividad residual del LDLR (nula, deficiente o indeterminada) se detallan en en el apartado «Métodos» y la tabla 1 del material adicional.
Tabla 1. Características de los pacientes con hipercolesterolemia familiar heterocigótica del estudio, agrupados por enfermedad cardiovascular prematura
| Sin ECVp (n=262) | Con ECVp (n=77) | p | |
| Varones | 111 (42,4) | 47 (35,9) | 0,004 |
| Mujeres | 151 (57,6) | 30 (41,1) | |
| Edad (años) | 45,2±15,3 | 53,19±11,3 | < 0,0001 |
| IMC | 26,3±4,8 | 28,01±5 | 0,014 |
| Hipertensión | 42 (16,1) | 20 (26) | 0,06 |
| Fumadores | 115 (43,8) | 42 (54,5) | 0,067 |
| Diabetes | 11 (4) | 3 (4) | 0,98 |
| Colesterol total (mg/dl) | 287,1±62,1 | 281,1±70,2 | 0,512 |
| cLDL (mg/dl) | 215,3±58,7 | 209±67,7 | 0,925 |
| cHDL (mg/dl) | 52,2±13,0 | 51,3±17,4 | 0,24 |
| Triglicéridos (mg/dl) | 92,5 (38-320) | 94,5 (44-258) | 0,589 |
| ApoA (mg/dl) | 141,1±35,2 | 138,8±32,1 | 0,204 |
| ApoB (mg/dl) | 166,4±43,9 | 159,4±41,0 | 0,256 |
| Efecto de la mutación en la actividad del LDLR | 0,527 | ||
| Alelo nulo | 161 (61,6) | 51 (66,2) | |
| Actividad deficiente | 75 (28,5) | 22 (28,6) | |
| Actividad indeterminada | 26 (9,9) | 4 (5,2) |
ApoA: apolipoproteína A; ApoB: apolipoproteína B; cHDL: colesterol unido a lipoproteínas de alta densidad; cLDL: colesterol unido a lipoproteínas de baja densidad; ECVp: enfermedad cardiovascular prematura; IMC: índice de masa corporal; LDLR: receptor de lipoproteínas de baja densidad.
Los resultados se muestran como media±desviación estándar, n (%) o mediana (intervalo).
Se consideraron estadísticamente significativos los valores de p<0,05.
En primer lugar, se realizó una búsqueda de polimorfismos en el promotor del LRP1 mediante polimorfismos de conformación de cadena sencilla del ADN y secuenciación en una muestra de 86 individuos con HFh, y en segundo lugar se analizó la asociación genética de un total de 10 polimorfismos con ECVp. El ADN se extrajo de sangre periférica con el equipo comercial Wizard® Genomic DNA Purification de Promega.
Cribado de polimorfismos del promotor del LRP1Véase «Métodos» y tabla 2 del material adicional.
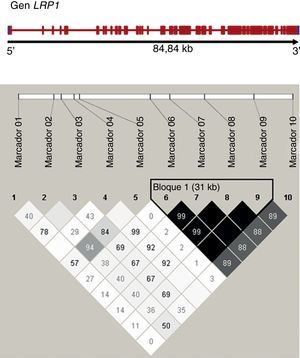
Estudio de asociación genotípicaSe seleccionó un total de 10 polimorfismos de un solo nucleótido (SNP) del LRP1, junto con el polimorfismo hallado en el promotor, en función de su frecuencia, referencias bibliográficas y funcionalidad (Tabla 2 y Figura). Los polimorfismos se genotipificaron mediante TaqMan®-reacción en cadena de la polimerasa y discriminación alélica en un aparato Applied Biosystem 7900.
Tabla 2. Características de los polimorfismos analizados
| Polimorfismo analizado | Variantes genómicas | SNP ID dbSNP | Alelos 1>2 a | Localización génica | Efecto funcional | MAF publicada b | MAF en este estudio |
| 1 | c.1-25C>G | rs35282763 | C>G | Promotor | Nueva secuencia Sp1 | 0,08 | 0,08 |
| 2 | IVS2+617C>T | rs715948 | C>T | Intrón 2 | — | 0,32 | 0,31 |
| 3 | c.677C>T | rs1799986 | C>T | Exón 3 | Asp100Asp | 0,14-0,15 | 0,14 |
| 4 | c.1116C>T | rs1800127 | C>T | Exón 6 | Ala217Val | 0,03 | 0,03 |
| 5 | IVS6+245C>G | rs7968719 | G>C | Intrón 6 | — | 0,49 | 0,47 |
| 6 | IVS19+144C>T | rs1800176 | C>T | Intrón 19 | — | 0,30 | 0,30 |
| 7 | c.4012C>T | rs1800194 | C>T | Exón 22 | Cys1182Cys | 0,31 | 0,30 |
| 8 | IVS38+53C>T | rs1800181 | C>T | Intrón 38 | - | 0,30 | 0,30 |
| 9 | c.10249G>A | rs1140648 | G>A | Exón 61 | Thr3261Thr | 0,28 | 0,30 |
| 10 | IVS83+139A>G | rs1800164 | G>A | Intrón 83 | — | 0,28 | 0,35 |
dbSNP: single nucleotide polymorphism database; MAF: minor allele frequency; SNP ID: single nucleotide polymorphism identification.
a El alelo 1 corresponde al de mayor frecuencia.
b Frecuencia alélica menor de panel CEU o CEPH de HapMap, hallada en bibliografía 8,15,16 .
Figura. Análisis de desequilibrio de ligamento entre los polimorfismos de un solo nucleótido del LRP1 analizados. Bloques de haplotipos definidos según el método de Gabriel (Haploview 4.0), el desequilibrio de ligamento entre pares de marcadores se muestra como valores de r2 (en negro r2>0,8). Enmarcado en la derecha del diagrama, se muestra un bloque con fuerte ligamento formado por los polimorfismos de un solo nucleótido 6 a 9 del estudio.
Análisis funcional del polimorfismo c.677C>T (rs1799986)El posible efecto funcional del polimorfismo en el procesamiento del ARNm se analizó mediante soporte bioinformático y análisis in vitro de minigén. En dicha prueba, el exón de interés se clona entre dos exones sintéticos para el posterior análisis del procesamiento de ARN («Métodos» del material adicional).
Análisis estadísticoLos datos clínicos entre los grupos se compararon mediante la prueba de la t de Student para variables continuas, la de la χ2 para variables categóricas y el test no paramétrico para variables que no seguían una distribución normal (SPSS v.14.0). Las frecuencias alélicas se calcularon a partir de los genotipos de los sujetos. De cada polimorfismo, se comprobó que su distribución cumplía con la ley de Hardy-Weinberg mediante χ2. Para analizar la hipótesis de asociación entre los polimorfismos del gen LRP1 y ECVp en la HFh, se utilizaron tests de multivariables basados en regresión logística, al igual que para el análisis de interacción con covariables17, 18. Se calcularon las odds ratio (OR) y sus intervalos de confianza del 95% (IC95%) para cada genotipo comparado con el homocigoto del alelo más frecuente o de referencia, y se estimó la significación para un umbral de p<0,05 asumiendo un modelo de herencia general (tres genotipos por separado), y modelo dominante.
La potencia estadística previa de la muestra (77 casos y 262 controles) se estimó en un 80% para una prevalencia de la enfermedad del 20%, un efecto del polimorfismo en el riesgo de sufrir un evento cardiovascular > 2 y > 1,7 para frecuencias alélicas de 0,14 y 0,30 respectivamente (Quanto v 1.2). Se incluyeron marcadores con frecuencias alélicas inferiores por su funcionalidad (polimorfismo del promotor, SNP1, y mutación de cambio de aminoácido, SNP4).
Todos los análisis se ajustaron por edad, sexo y otros factores de riesgo de ECV en la población estudiada (Tabla 1). Se estimaron las frecuencias haplotípicas mediante desequilibrio de ligamiento (LD) en bloques definidos por el método de Gabriel, se calcularon los valores de LD y r2 entre pares de SNP mediante Haploview v. 4.2 (Figura).
ResultadosCaracterísticas de la población estudiada agrupada por enfermedad cardiovascular prematuraLas características de dicha población agrupada por la presencia de ECVp se muestran en la Tabla 1. El 86,5% de los pacientes con HFh se hallaba en tratamiento con terapia antilipemiante en el momento de la inclusión en el estudio. La enfermedad coronaria o cardiopatía isquémica representó el 93,4% de los eventos. Se observó mayor predisposición a padecer ECVp en los varones (p=0,004), en función de la edad (p<0,001) y con el aumento del índice de masa corporal (IMC) (p=0,01). Respecto al tabaquismo, la proporción de fumadores en el momento de inclusión no era significativamente diferente.
Se observaron diferencias entre sexos (datos no mostrados en la Tabla 1). El IMC fue 28,4±6,6 en mujeres con ECVp frente a 26,7±5,7 en mujeres sin ECVp (p=0,137) y 27,7±3,7 en varones con ECVp frente 25,9±3,1 en varones sin ECVp (p=0,003). Las frecuencias observadas para la hipertensión y el tabaquismo no fueron significativamente distintas entre ambos grupos, aunque la hipertensión fue más prevalente en mujeres (con ECVp frente a sin ECV, el 40 frente al 18,2%; p=0,014) que en varones (el 16,3 frente al 13,2%; p=0,612), y el tabaquismo fue más prevalente en varones (con ECVp frente a sin ECVp, el 82,6 frente al 58,7%; p=0,005) que en mujeres (el 16,7 frente al 33,7%; p=0,085). Si bien el porcentaje de individuos con ECVp fue superior entre los pacientes con HFh y mutaciones del LDLR de alelo nulo o de mayor gravedad, las diferencias no alcanzaron significación estadística en esta muestra.
Cribado de polimorfismos del promotor del LRP1Se halló un polimorfismo en el promotor, c.1-25C>G (rs35282763) (fig. 1A del material adicional) que creaba un nuevo sitio de unión de factores de transcripción Sp1 según el análisis in silico (fig. 1B del material adicional).
Asociación de polimorfismos y haplotipos con enfermedad cardiovascular prematura en hipercolesterolemia familiar heterocigóticaLa distribución de los genotipos se ajustó al equilibrio de Hardy-Weinberg en todos los polimorfismos analizados (p>0,2). Las frecuencias alélicas observadas en la población no difirieron significativamente de las ya publicadas (Tabla 2). Además, SNPs 2, 3, 5 y 6 son los marcadores más representativos de haplotipo según el HapMap 3 Genome Browser release#2.
Los resultados de la asociación con ECVp tras ajustar por edad, sexo, IMC y el efecto de la mutación del LDLR se detallan en la Tabla 3. Únicamente el SNP3 (rs1799986) mostró una asociación significativa con la ECVp con el modelo dominante (CT+TT frente a CC, OR=1,94; IC95%, 1,08-3,48; p=0,029). Tras ampliar la muestra a 648 pacientes con HFh (tabla 3 del material adicional) de los cuales 133 con ECVp y 515 sin ECVp y reanalizar los datos, se obtuvieron resultados similares (OR=1,83; IC95%, 1,16-2,88; p=0,011).
Tabla 3. Asociación de polimorfismos y enfermedad cardiovascular prematura en hipercolesterolemia familiar heterocigótica
| SNP | SNP ID | HFh (n=339) | Frecuencias genotípicas | pOR (IC95%) | ||
| 1 | rs35282763 | CC | CG | GG | 0,9 | |
| Sin ECVp | 0,84 | 0,15 | 0,04 | |||
| Con ECVp | 0,88 | 0,11 | 0,01 | |||
| 2 | rs715948 | CC | CT | TT | 0,86 | |
| Sin ECVp | 0,5 | 0,38 | 0,13 | |||
| Con ECVp | 0,51 | 0,39 | 0,1 | |||
| 3 | rs1799986 | CC | CT | TT | 0,029 | |
| Sin ECVp | 0,77 | 0,2 | 0,02 | 1,94 (1,08-3,48) | ||
| Con ECVp | 0,64 | 0,36 | 0 | CT+TT frente a CC | ||
| 3 * | rs1799986 | CC | CT | TT | 0,011 | |
| Sin ECVp | 0,76 | 0,22 | 0,02 | 1,83 (1,16-2,88) | ||
| Con ECVp | 0,66 | 0,34 | 0 | CT+TT frente a CC | ||
| 4 | rs1800127 | CC | CT | TT | NS | |
| Sin ECVp | 0,95 | 0,05 | 0 | |||
| Con ECVp | 1 | 0 | 0 | |||
| 5 | rs7968719 | CC | CG | GG | 0,79 | |
| Sin ECVp | 0,29 | 0,47 | 0,24 | |||
| Con ECVp | 0,33 | 0,39 | 0,28 | |||
| 6 | rs1800176 | CC | CT | TT | 0,096 | |
| Sin ECVp | 0,46 | 0,45 | 0,09 | |||
| Con ECVp | 0,48 | 0,49 | 0,03 | |||
| 7 | rs1800194 | CC | CT | TT | 0,07 | |
| Sin ECVp | 0,47 | 0,43 | 0,1 | |||
| Con ECVp | 0,48 | 0,49 | 0,03 | |||
| 8 | rs1800181 | CC | CT | TT | 0,07 | |
| Sin ECVp | 0,47 | 0,43 | 0,1 | |||
| Con ECVp | 0,48 | 0,49 | 0,03 | |||
| 9 | rs1140648 | CC | CT | TT | 0,075 | |
| Sin ECVp | 0,46 | 0,45 | 0,1 | |||
| Con ECVp | 0,48 | 0,49 | 0,03 | |||
| 10 | rs1800164 | GG | AG | AA | 0,72 | |
| Sin ECVp | 0,42 | 0,45 | 0,13 | |||
| Con ECVp | 0,40 | 0,48 | 0,12 | |||
ECVp: enfermedad cardiovascular prematura; HFh: hipercolesterolemia familiar heterocigótica; IC95%: intervalo de confianza del 95%; ID: identificación; NS: no significativo; OR: odds ratio; SNP: single nucleotide polymorphism.
Todos los valores se han ajustado por edad, sexo, índice de masa corporal y tipo de mutación del receptor de lipoproteínas de baja densidad; rs1799986 muestra un valor de p significativo (modelo dominante, genotipos CT+TT frente a CC).
* Resultados tras aumentar la muestra con 515 sujetos sin ECVp y 133 con ECVp.
La frecuencia alélica para el polimorfismo rs1799986 en la poblacion con HF fue similar a la de poblacion de control española (p=0,76) y otras poblaciones caucásicas («Métodos» y tabla 4 del material adicional).
Se analizó la interacción del polimorfismo rs1799986 con el tipo de mutación del LDLR (de alelo nulo, actividad deficiente o indeterminada), y se observó un porcentaje superior de mutación de alelo nulo en individuos con el polimorfismo y ECVp respecto a los pacientes sin ECVp, pero el tamaño de la muestra no permite extraer resultados concluyentes (tabla 5 del material adicional).
Mediante Haploview, se examinó la existencia de bloques de polimofismos con fuerte desequilibrio de ligamento. Se observó únicamente un bloque formado por los SNP 6 a 9, según el método de Gabriel (Figura), sin que ningún haplotipo se asociara con la enfermedad (Tabla 4).
Tabla 4. Asociación de haplotipos con enfermedad cardiovascular prematura, ajustados por factores riesgo (n=339)
| Frecuencia de haplotipos | OR (IC95%) para el más frecuente | p | |
| CCCA | 0,692 (0,727-0,682) | 1 | |
| TTTG | 0,298 (0,306-0,293) | 0,84 (0,54-1,31) | 0,44 |
| Asociación general para haplotipos | 0,15 |
IC95%: intervalo de confianza del 95%; OR: odds ratio.
Frecuencia de los haplotipos del bloque con fuerte desequilibrio de ligamento, LD, ligamento formado por los polimorfismos de un solo nucleótido 6 a 9, estimada por el algoritmo de máxima expectación; frecuencia total (con enfermedad cardiovascular prematura/sin enfermedad cardiovascular prematura).
Como ya se ha comentado en el apartado «Análisis estadístico», el tamaño muestral inicial del estudio sólo permitía detectar asociaciones con OR>2, con potencia del 80% para frecuencias alélicas de 0,14 como la que presenta el SNP 3. Dado que el estudio de cohorte ha seguido incorporando nuevos pacientes, se pudo ampliar el tamaño muestral de individuos con HFh a un total de 133 con ECVp y 515 sin ella (tabla 3 del material adicional) para detectar como estadísticamente significativa una OR=1,83 con el 80% de potencia estadística para esta frecuencia alélica (Quanto v1.2.0).
Los resultados parecen indicar que la presencia del alelo T del polimorfismo c.677C>T (rs1799986) se asocia con un aumento del riesgo de sufrir ECVp en la HFh.
Análisis funcional del polimorfismo c.677C>T (rs179998)Los resultados del estudio funcional descartan que dicho polimorfismo pueda alterar el patrón normal de procesamiento del ARNm («Resultados» y fig. 2 del material adicional).
DiscusiónEstudios anteriores han citado la asociación de polimorfismos del LRP1 con enfermedad coronaria en poblaciones caucásicas8, 19, 20 con enfermedad coronaria y longevidad21 y con trombosis22, 23, aunque otros estudios han presentado resultados menos significativos o contradictorios para dicha asociación en relación con la dislipemia o la enfermedad de Alzheimer15, y también en relación con la trombosis24. Estos y otros trabajos publicados sobre la implicación de los polimorfismos del LRP1 en enfermedades complejas se recogen en el resumen de Gläser et al16. Recientemente, Peters et al25, en un amplio estudio de SNP de genes candidatos implicados en la eficacia de las estatinas en la disminución del colesterol, hallaron que un polimorfismo del intrón 2 del LRP1 (rs715948, SNP 2 del presente estudio) se asociaba con una disminución de infarto de miocardio, lo que no se ha replicado en nuestro estudio de HFh con ECV, fenotipo más amplio que el citado anteriormente.
Se han publicado resultados contradictorios respecto a la implicación del polimorfismo c.677C>T con ECV y población caucásica. Pocathikorn et al20, en un estudio con 600 individuos con enfermedad coronaria y 700 controles, hallaron una frecuencia significativamente inferior del genotipo TT del polimorfismo c.677C>T en individuos con enfermedad coronaria respecto a los controles; por el contrario, Benes et al21, con 654 individuos con enfermedad coronaria y 525 controles, hallaron que la presencia del alelo T aumentaba el riesgo de padecer enfermedad coronaria en los sujetos con el genotipo 5G/5G del inhibidor del activador del plaminógeno tisular de tipo 1.
Aunque en el presente estudio se ha intentado analizar polimorfismos a lo largo de todo el gen, dado su tamaño de aproximadamente 85 kb, sería necesario un mayor número de SNP para cubrir el máximo de variabilidad en la región, y principalmente en el extremo 5’, donde hay menor densidad de polimorfismos con LD según el análisis de la región chr12:5508548..55893389 (HapMap3 Genome Browser release #2) con Haploview (resultados no mostrados).
LimitacionesEntre las limitaciones del estudio cabe reseñar, además de las derivadas del tamaño de la muestra y la potencia del ensayo, las que se originan en el número de polimorfismos analizados y la probabilidad de encontrar una asociación por azar. Así pues, los resultados presentados deberían repetirse en otras poblaciones para poder confirmarlos y proceder posteriormente a la búsqueda de otros polimorfismos en LD con la variante genética asociada al riesgo de sufrir ECVp.
El estudio diseñado computa los eventos cardiovasculares que han aparecido con anterioridad a la inclusión en el estudio y según las pautas de edad establecidas para ECVp; sin embargo, al ser una cohorte retrospectiva, puede existir un sesgo de selección, ya que algunos casos mortales no se han incluido en el estudio. Una limitación importante del diseño del estudio estriba en que el fenotipo que se analiza es a punto final, evento cardiovascular, por lo que carecemos de datos sobre el grado de afección aterosclerótica previa al evento, como podría ser número de vasos afectados de aterosclerosis; ello requiere otro tipo de diseño y análisis clínicos no considerados por ahora en la totalidad de las muestras.
Como se ha descrito en la literatura médica26, el efecto aditivo de diversas variantes genéticas puede ayudar a definir mejor el riesgo de sufrir cardiopatía isquémica y, por extensión, ECV. Por ello sería interesante el análisis de variantes funcionales del LRP1 junto con otros genes asociados con la enfermedad y/o implicados en procesos similares.
ConclusionesParece que la presencia del alelo T del polimorfismo c.677C>T (rs179998) confiere mayor riesgo de ECVp en la HFh. Dado que la funcionalidad del polimorfismo no se ha demostrado, cabe suponer que este se halla en LD con otra variante genética que causa el aumento del riesgo de ECV. Son necesarios más estudios con otras poblaciones para replicar los resultados y, en caso afirmativo, descubrir qué polimorfismo(s) se halla(n) en estrecho LD con rs179998 y en asociación con la ECV.
FinanciaciónEste trabajo se ha realizado en parte gracias a la financiación del SAF2010-16549, CNIC-08-2008, CIBERobn CB06/03.
Conflicto de interesesNinguno.
Agradecimientos
A Nuria Sala, Xavier Muñoz (ICO-IDIBELL) y Mónica Gratacós (CRG), por su discusión científica. A Montse Gómez por su asistencia técnica y a la Fundación de Hipercolesterolemia Familiar por su soporte al estudio.
Appendix A. Material adicionalSe puede consultar material adicional a este artículo en su versión electrónica disponible en http://dx.doi.org/10.1016/j.recesp.2012.03.013.
Material suplementarioRecibido 6 Octubre 2011
Aceptado 7 Marzo 2012
Autor para correspondencia: Centro de Investigación Cardiovascular, CSIC-ICCC, Hospital de la Santa Creu i Sant Pau, Sant Antoni Maria Claret 167, 08025 Barcelona, España. lbadimon@csic-iccc.org