SEC 2021 - El Congreso de la Salud Cardiovascular
Introducción
Dr. Héctor Bueno
Presidente del Comité Científico del Congreso
Comité ejecutivo
Comité de evaluadores
Listado de sesiones
Índice de autores
4017. Nuevos datos epidemiológicos, innovaciones tecnológicas y avances científicos en el área cardiovascular
4017-2. IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
Hospital de Bellvitge, Hospitalet de Llobregat, Barcelona.
Introducción y objetivos: El déficit de hierro (DH) es una entidad frecuente en la insuficiencia cardiaca (IC), observándose en aproximadamente la mitad de los pacientes y asociándose con peor pronóstico y calidad de vida. El objetivo del estudio fue identificar patrones de expresión genética en sangre periférica asociados con el metabolismo del DH y que pudieran estar implicados en la fisiopatología de la IC.
Métodos: Se analizaron datos a partir de muestras de sangre periférica del estudio DAMOCLES (Definition of the neurohormonal Activation, Myocardial function, genOmic expression an CLinical outcomes in hEart failure patientS), una cohorte prospectiva unicéntrica de 1,236 pacientes con IC; considerándose en este proyecto pacientes con FEVI 12 g/dl). Se seleccionaron catorce pacientes para un análisis completo del transcriptoma (RNAm), 7 con DH y 7 sin DH (grupo control), con el objetivo de identificar expresión genética diferencial entre ambos grupos. A partir de este punto, se realiza una selección de genes sobreexpresados en el grupo de DH tras ajustar por covariables y basándose en evidencia científica previa.
Resultados: Excluyendo la definición de DH, no había otras diferencias entre los dos grupos analizado. Se apreciaron 1.128 transcripciones sobreexpresadas en el grupo de DH respecto al grupo control. Tras ajustar inicialmente por covariables, basándonos en la importancia clínica y focalizándonos en los genes asociados a vías metabólicas que están relacionadas con la IC, la regulación y el metabolismo del hierro celular, se seleccionaron 22 genes para análisis adicionales. A continuación realizamos un exámenes exhaustivos de ontología genética y se seleccionaron finalmente 6 genes, que resultaron ser componentes bien conocidos del eje neurohormonal de la IC, la homeostasis del hierro y el metabolismo mitocondrial: receptor de transferrina (TFRC), ferritina mitocondrial (FTMT), sirtuina 7 (SIRT7), pequeña proteína integral de membrana 20 (SMIM20), adrenomedulina (ADM) y enzima convertidora de endotelina 1 (ECE1).
|
Resultados del análisis TLDA |
||
|
Símbolo |
ID vs Ct |
p |
|
ACO1 |
0,76 |
0,18 |
|
ADM |
0,62 |
0,03 |
|
COA3 |
0,85 |
0,42 |
|
COX17 |
0,66 |
0,08 |
|
CXCL8 |
1,18 |
0,53 |
|
ECE1 |
0,70 |
0,03 |
|
FTMT |
0,26 |
0,02 |
|
HADHA |
0,95 |
0,74 |
|
LCN2 |
0,86 |
0,36 |
|
LIG3 |
0,71 |
0,09 |
|
MCUR1 |
0,76 |
0,15 |
|
MT2A |
1,05 |
0,79 |
|
MYH7B |
0,87 |
0,56 |
|
SIRT7 |
0,63 |
0,02 |
|
SMIM20 |
0,68 |
0,04 |
|
STEAP3 |
1,02 |
0,92 |
|
TFRC |
1,31 |
0,03 |
|
YME1L1 |
0,82 |
0,28 |
|
ZNF260 |
0,51 |
0,13 |
|
Fold change y valor p de los genes analizados por TLDA. |
||
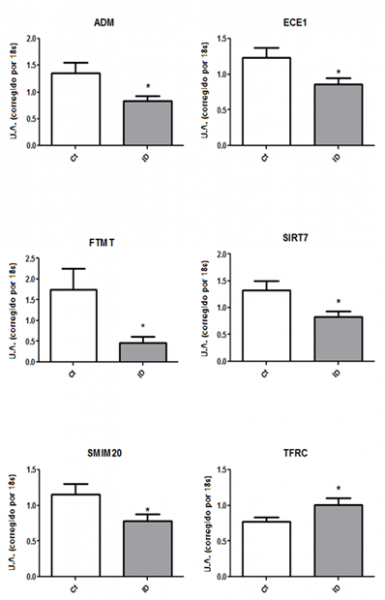
Representación gráfica de los 6 genes expresados de forma diferenciada. Análisis de los niveles de ARNm de ADM, ECE1, FTMT, SIRT7, SMIM20 y TFRC. Los datos fueron normalizados y expresados como media ± SEM. (*p < 0,05 vs Control).
Conclusiones: En nuestra cohorte de pacientes con IC se identificaron 6 genes con expresión diferenciada en el grupo con DH, todos implicados en vías de la homeostasis del hierro y eje neurohormonal de la IC. Nuestro trabajo permite realizar hipótesis de que estas vías génicas podrían estar compartidas en la fisiopatología tanto de la IC como del DH.
Comunicaciones disponibles de "Nuevos datos epidemiológicos, innovaciones tecnológicas y avances científicos en el área cardiovascular"
- 4017-1. MODERADOR
- Antoni Bayés Genís, Barcelona
- 4017-2. IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- Santiago Jiménez Marrero, Lidia Alcoberro Torres, Marta Tajes Orduña, Sergi Yun Viladomat, Alberto Garay Melero, Pedro Moliner Borja, Cristina Enjuanes Grau, Esther Calero Molina, Encarna Hidalgo Quirós, Lydia Sánchez Fernández, Nuria Juan, Marta Ruiz, María del Carmen Basalo Carbajales, Josep Comin-Colet y Carles Díez
Hospital de Bellvitge, Hospitalet de Llobregat, Barcelona.
- 4017-3. EFICACIA Y SEGURIDAD DE UNA CONSULTA ELECTRÓNICA (E-CONSULTA) EN LOS PACIENTES CON ENFERMEDAD CARDIOVASCULAR
- David García Vega1, Belén Álvarez Álvarez1, Pilar Mazón Ramos1, Ricardo Lage Fernández1, Diego Iglesias Álvarez1, Francisco Gude Sampedro1, Daniel Rey Aldana2, Manuel Portela-Romero3, Sergio Cinza-Sanjurjo4 y José Ramón González-Juanatey1
1Complexo Hospitalario Universitario de Santiago de Compostela, A Coruña. 2Centro de Salud A Estrada, Pontevedra. 3Centro de Salud Concepción Arenal, Santiago de Compostela, A Coruña. 4Centro de Salud de Porto do Son, A Coruña.
- 4017-4. EVOLUCIÓN PARADÓJICA DE LA LA TASA DE MUERTE POR INSUFICIENCIA CARDIACA (TMIC) EN ESPAÑA EN LA ÚLTIMA DÉCADA
- Lucía Fernández-Bobadilla1, María G. Crespo-Leiro2, Juan Ramón Rey-Blas3, Eduardo Barge-Caballero2, Esteban López de Sá y Areses3, Gonzalo Barge-Caballero2 y Jaime Fernández de Bobadilla Osorio3
1Universidad de Santiago de Compostela, A Coruña. 2Complexo Hospitalario Universitario A Coruña. 3Hospital Universitario La Paz, Madrid.
- 4017-5. INFLUENCIA DEL ÁREA DE SALUD DE RESIDENCIA EN EL MÉTODO DE REPERFUSIÓN PRIMARIA Y MORTALIDAD EN LOS PACIENTES CON INFARTO AGUDO DE MIOCARDIO CON ELEVACIÓN DEL ST EN LA COMUNIDAD FORAL DE NAVARRA, AÑOS 2017-2019
- Ignacio Roy Añón1, Nerea Mora Ayestarán1, Marina Oliver Ledesma1, M. Soledad Alcasena Juango1, Juan José Gavira Gómez2, Irene Iniesta Martínez3 y Conchi Moreno Iribas3
1Complejo Hospitalario de Navarra, Pamplona/Iruña, Navarra. 2Clínica Universidad de Navarra, Pamplona/Iruña, Navarra. 3Instituto de Salud Pública y Laboral de Navarra, Pamplona/Iruña, Navarra.
- 4017-6. TRANSFERENCIA DE APRENDIZAJE PARA LA SEGMENTACIÓN AUTOMÁTICA 3D DE LA AORTA A PARTIR DE ANGIOGRAFÍA POR RESONANCIA MAGNÉTICA SIN CONTRASTE
- Andrea Guala1, Jordina Avilés2, Gonzalo D. Maso Talou2, Óscar Camara2, Marcos Mejía Córdova2, Edward Ferdian2, Kat Gilbert2, Alistar A. Young3, Lydia Dux-Santoy Hurtado1, Aroa Ruiz Muñoz1, Gisela Teixido-Tura4 y José Rodríguez-Palomares4
1Vall d'Hebron Institut de Recerca, Barcelona. 2Universitat Pompeu Fabra, Barcelona. 3King's College London, Londres (Reino Unido). 4Hospital Universitari Vall d'Hebron, Barcelona.
- 4017-7. CAMBIOS EN EL LIPIDOMA DE LOS DEPÓSITOS VISCERAL Y SUBCUTÁNEO DEL TEJIDO ADIPOSO INDUCIDOS POR EL TRATAMIENTO CON EMPAGLIFLOZINA EN RATAS OBESAS DIABÉTICAS ZUCKER
- Laura Anido Varela1, Sandra Moraña Fernández2, Alana Aragón Herrera3, Sandra Feijóo Bandín3, Manuel Otero Santiago4, Estefanía Tarazón5, Esther Roselló Lleti5, Manolo Portolés5, Manuel Campos Toimil6, José Ramón González Juanatey7 y Francisca Lago Paz7
1Instituto de Investigación Sanitaria de Santiago de Compostela, A Coruña. 2Centro Singular de Investigación en Medicina Molecular y Enfermedades Crónicas, Instituto de Investigación Sanitaria de Santiago de Compostela, A Coruña. 3Instituto de Investigación Sanitaria de Santiago de Compostela, Complexo Hospitalario Universitario de Santiago de Compostela, A Coruña. 4Instituto de Investigación Sanitaria de Santiago de Compostela, Santiago de Compostela, A Coruña. 5Hospital Universitari i Politècnic La Fe, Valencia. 6Physiology and Pharmacology of Chronic Diseases, Center for Research in Molecular Medicine and Chronic Diseases, Universidade de Santiago de Compostela, A Coruña. 7Complexo Hospitalario Universitario de Santiago de Compostela, A Coruña.
Más comunicaciones de los autores
-
Alcoberro Torres, Lidia
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6003-10 - TRATAMIENTO ANTITROMBÓTICO EN LAS DISECCIONES CORONARIAS ESPONTÁNEAS Y SU IMPACTO A NIVEL DE EVENTOS CLÍNICOS DURANTE EL INGRESO
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
-
Basalo Carbajales, María del Carmen
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
-
Calero Molina, Esther
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
-
Comin-Colet, Josep
- 6029-7 - IMPACTO DEL USO DE TERAPIA ANTIAGREGANTE CRÓNICA EN EL PRONÓSTICO DE PACIENTES CON DISECCIÓN CORONARIA ESPONTÁNEA
- 5029-5 - LA GAMMAGRAFÍA CON DPD NO DETECTA LA AFECTACIÓN CARDIACA POR AMILOIDOSIS TRANSTIRRETINA EN ENFERMEDAD DE INICIO PRECOZ ATTRV30M
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5004-5 - IMPACTO SOBRE LA AGREGABILIDAD PLAQUETARIA DE LA INFECCIÓN POR CORONAVIRUS SARS-COV-2
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 6003-10 - TRATAMIENTO ANTITROMBÓTICO EN LAS DISECCIONES CORONARIAS ESPONTÁNEAS Y SU IMPACTO A NIVEL DE EVENTOS CLÍNICOS DURANTE EL INGRESO
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
-
Díez López, Carles
- 5029-5 - LA GAMMAGRAFÍA CON DPD NO DETECTA LA AFECTACIÓN CARDIACA POR AMILOIDOSIS TRANSTIRRETINA EN ENFERMEDAD DE INICIO PRECOZ ATTRV30M
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 5029-1 - MODERADOR
-
Enjuanes Grau, Cristina
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
-
Garay Melero, Alberto
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
-
Hidalgo Quirós, Encarnació
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
-
Jiménez Marrero, Santiago
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
-
Juan, Nuria
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
-
Moliner Borja, Pedro
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
-
Ruiz, Marta
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
-
Sánchez Fernández, Lydia
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
-
Tajes Orduña, Marta
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
-
Yun Viladomat, Sergi
- 6008-4 - DETERMINANTES FÍSICOS DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 6039-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA Y SU ASOCIACIÓN CON HOSPITALIZACIÓN Y MORTALIDAD
- 5007-8 - EFICACIA DE LA TELEMEDICINA EN PACIENTES CON INSUFICIENCIA CARDIACA EN FUNCIÓN DEL FENOTIPO DE FRAGILIDAD EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DEL ESTUDIO ICOR (INSUFICIÈNCIA CARDIACA OPTIMITZACIÓ REMOTA)
- 5029-5 - LA GAMMAGRAFÍA CON DPD NO DETECTA LA AFECTACIÓN CARDIACA POR AMILOIDOSIS TRANSTIRRETINA EN ENFERMEDAD DE INICIO PRECOZ ATTRV30M
- 5007-6 - FENOTIPOS DE FRAGILIDAD EN PACIENTES CON INSUFICIENCIA CARDIACA EN EL PERIODO TRANSICIONAL POSALTA: SUBANÁLISIS DE CLUSTERS BASADO EN MACHINE LEARNING DEL ENSAYO CLÍNICO ICOR
- 6008-5 - DETERMINANTES PSICOSOCIALES DE MALA CALIDAD DE VIDA GLOBAL Y SUS DIMENSIONES EN INSUFICIENCIA CARDIACA CRÓNICA: ANÁLISIS DE 1.236 PACIENTES EN VIDA REAL
- 4017-2 - IDENTIFICACIÓN DE VÍAS GENÉTICAS ASOCIADAS AL DÉFICIT DE HIERRO A PARTIR DE MUESTRAS DE TRANSCRIPTOMA EN SANGRE PERIFÉRICA EN PACIENTES CON INSUFICIENCIA CARDIACA
