SEC 2024 - El Congreso de la Salud Cardiovascular
Introducción
Dr. José María de la Torre Hernández
Presidente del Comité Científico del Congreso. Vicepresidente de la SEC
Comités ejecutivo, organizador y científico
Comité de evaluadores
Listado completo de comunicaciones
Índice de autores
4008. Comunicaciones en cardiogenética, miocardiopatías y aortopatías
4008-3. Impacto de variantes sinónimas en las miocardiopatías hereditarias
1Cardiología. Centro de Investigación Biomédica en Red de Enfermedades Cardiovasculares (CIBER-CV), Madrid, España, 2Cardiología. Hospital General Universitario Gregorio Marañón, Madrid, España, 3Cardiología. Hospital Universitario de Getafe, Getafe (Madrid), España y 4Cardiología Pediátrica. Hospital General Universitario Gregorio Marañón, Madrid, España.
Introducción y objetivos: Las variantes sinónimas en miocardiopatías hereditarias suelen clasificarse como benignas al no producir un cambio directo en la secuencia de aminoácidos. Pero un cambio de nucleótido puede repercutir en la estructura y funcionalidad de la proteína por diferentes mecanismos. De hecho, variantes sinónimas ya han ido reclasificadas como patogénicas. Existen nuevas herramientas de análisis in silico que permiten determinar el potencial impacto de estas variantes y reevaluar su papel causal. El objetivo del estudio fue evaluar estas variantes en una cohorte de pacientes con miocardiopatía.
Métodos: Se revisaron retrospectivamente los análisis genéticos (exoma clínico o exoma completo) de 1.500 probandos de una Unidad Cardiopatías Familiares (marzo de 2019-febrero de 2024). Se analizaron específicamente variantes sinónimas, su frecuencia y potencial impacto (herramientas synVEP, usDSM, RNAfold, SpliceAI), en genes principales de miocardiopatías con herencia autosómica dominante (30 genes).
Resultados: Se detectaron 1.172 variantes sinónimas diferentes en 1500 pacientes. Asumiendo que las variantes neutras surgen de manera más frecuente que las deletéreas, se determinó el número de variantes sinónimas, a priori consideradas neutras, y se comparó con el número de variantes missense (figura). La aparición de variantes sinónimas en los genes LMNA, DES, MYL2, CSRP3 y TPM1 fue dos veces superior a la de missense, indicando neutralidad, mientras que para el resto de genes fue igual o inferior que las missense, incluso 3 veces inferior para PKP2, MYBPC3, ACTC1 y TNNC1, indicando no neutralidad. De las 1.172 variantes sinónimas, 27 en 29 pacientes (5 con miocardiopatía arritmogénica, 14 con miocardiopatía dilatada, 9 con miocardiopatía hipertrófica y 1 con muerte súbita no aclarada, 23 de ellos con genética negativa) estaban ausentes en población general y tenían potencial impacto deletéreo. Por tanto, se reclasificaron y pasaron a ser consideradas candidatas a determinar el fenotipo de estos pacientes (tabla).
|
Variantes sinónimas candidatas |
|||||||||
|
Gen |
cDNA |
Frecuencia (%) |
synVEP |
usDSM |
Splicing AI |
RNAfold |
Paciente |
Fenotipo |
Otra variante (P/LP) |
|
MYH7 |
c.2265G> A |
0,001 |
0,763 |
0,714 |
0,000 |
S,E |
1 |
MCD |
No |
|
c.4948T> C |
0,001 |
0,686 |
0,611 |
0,000 |
S,E |
2 |
MCD |
No |
|
|
c.4618C> T |
0,007 |
0,676 |
0,706 |
0,000 |
S,E |
3 |
MCD |
No |
|
|
c.432C> A |
0,000 |
0,917 |
0,700 |
0,000 |
S,E |
4 |
MCD |
No |
|
|
c.3846C> A |
0,000 |
0,622 |
0,721 |
0,000 |
No |
5 |
MCH |
MYH7 |
|
|
FLNC |
c.126C> T |
0,000 |
0,634 |
0,774 |
0,000 |
No |
6 |
MCD |
No |
|
c.3090C> T |
0,000 |
0,506 |
0,168 |
0,020 |
No |
7 |
MCH |
No |
|
|
c.531C> T |
0,000 |
0,684 |
0,512 |
0,000 |
No |
8 |
MCD |
No |
|
|
TNNT2 |
c.255G> T |
0,000 |
0,993 |
0,82 |
0,010 |
S,E |
9 |
MCD |
No |
|
c.453G> C |
0,000 |
0,93 |
0,506 |
0,000 |
S,E |
10 |
MCD |
No |
|
|
MYH6 |
c.4560A> G |
0,006 |
0,601 |
0,684 |
0,030 |
No |
11 |
MCH |
MYL3 |
|
c.3609C> G |
0,001 |
0,527 |
0,679 |
0,000 |
No |
12 |
MCD |
MYH7 |
|
|
c.3609C> G |
0,001 |
0,527 |
0,679 |
0,000 |
No |
13 |
MCH |
SOS1 |
|
|
c.3609C> G |
0,001 |
0,527 |
0,679 |
0,000 |
No |
14 |
MCH |
no |
|
|
c.4419G> A |
0,000 |
0,745 |
0,839 |
0,030 |
E |
15 |
MCH |
no |
|
|
c.732C> T |
0,001 |
0,779 |
0,750 |
0,000 |
No |
16 |
MCD |
LZTR1 |
|
|
c.546G> A |
0,003 |
0,703 |
0,587 |
0,020 |
No |
17 |
MCD |
SCN5A |
|
|
SCN5A |
c.1578C> A |
0,000 |
0,900 |
0,271 |
0,200 |
E |
18 |
MCA |
No |
|
BAG3 |
c.465A> G |
0,001 |
0,689 |
0,341 |
0,000 |
S,E |
19 |
MCA |
No |
|
c.456A> G |
0,001 |
0,887 |
0,449 |
0,000 |
S,E |
20 |
MCD |
No |
|
|
DSP |
c.81C> A |
0,001 |
0,907 |
0,476 |
0,000 |
S,E |
21 |
MCH |
No |
|
c.6471A> C |
0,006 |
0,932 |
0,202 |
0,000 |
S,E |
22 |
MCD |
No |
|
|
JUP |
c.72G> A |
0,000 |
0,894 |
0,200 |
0,000 |
E |
23 |
MCD |
No |
|
c.1815C> G |
0,000 |
0,807 |
0,303 |
0,000 |
S,E |
24 |
MCA |
No |
|
|
c.351C> T |
0,002 |
0,783 |
0,485 |
0,020 |
S,E |
25 |
MCH |
No |
|
|
c.468G> A |
0,000 |
0,817 |
0,982 |
0,830 |
S,E |
26 |
MCA-Naxos |
No |
|
|
TTR |
c.342A> T |
0,000 |
0,989 |
0,704 |
0,000 |
E |
27 |
MS |
No |
|
PKP2 |
c.1464C> T |
0,001 |
0,913 |
0,592 |
0,240 |
S,E |
27 |
MS |
No |
|
c.795G> T |
0,007 |
0,942 |
0,517 |
0,000 |
S,E |
28 |
MCA |
No |
|
|
RBM20 |
c.978G> A |
0,001 |
0,556 |
0,645 |
0,010 |
No |
29 |
MCH |
NF1 |
|
MCD: miocardiopatía dilatada; MCH: miocardiopatía hipertrófica; MCA: miocardiopatía arritmogénica; MS: muerte súbita; synVEP: predictor basado en aprendizaje automático para evaluar los efectos de variantes sinónimas humanas (0-1); usDSM: predictor basado en random forest para detectar mutaciones sinónimas nocivas (0-1); Splicing AI: predictor de splicing mediante inteligencia artificial (0-1); RNAfold: predictor de estructura secundaria del RNA (S: estructura, E: entropía). |
|||||||||
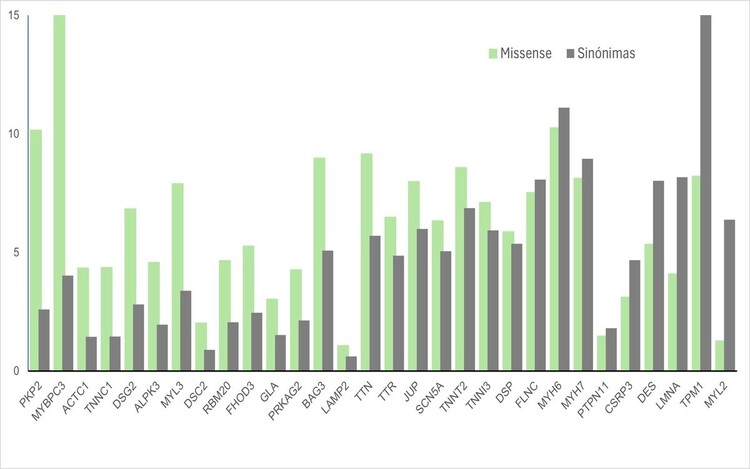
Ratio de variantes sinónimas y missense (número de variantes/tamaño transcrito en Kb) en los 30 genes analizados.
Conclusiones: Las nuevas herramientas de predicción nos han permitido reclasificar el 2% de las variantes sinónimas de benignas a variantes de significado incierto, candidatas a estudios de segregación fenotipo-genotipo. Las nuevas evidencias apoyan la necesidad de reanalizar este tipo de variantes con las nuevas herramientas de predicción disponibles y tenerlas en cuenta en los estudios genéticos.
Comunicaciones disponibles de "4008. Comunicaciones en cardiogenética, miocardiopatías y aortopatías"
- 4008-1. Modera
- Almudena Amor Salamanca. Área de Genética Cardiovascular, Health In Code (A Coruña)
- 4008-2. Caracterización de polimorfismos genéticos del CYP450 asociados a síndrome de QT largo inducido por fármacos en una gran cohorte prospectiva
- Bieito Campos García1, Juliana Salazar Blanco2, Benjamín Rodríguez Santiago3, Héctor Hernández Ontiveros4, Aina Ávila Parcet5, Víctor García Hernando1, Mar Carceller Sindreu5, Ana Juanes Borrego6, María Antonia Martí Femenías7, Enrique Rodríguez Font1, Concepción Alonso Martín1, Zoraida L. Moreno Weidmann1, Francisco Méndez Zurita1, Xavier Viñolas Prat1 y José M.M. Guerra Ramos1
1Servicio de Cardiología. Hospital de la Santa Creu i Sant Pau, Barcelona, España, 2Unidad de Genética. Institut d’Investigació Biomèdica Sant Pau, Barcelona, España, 3Servicio de Genética. Hospital de la Santa Creu i Sant Pau, Barcelona, España, 4Servicio de Medicina Interna. Hospital de la Santa Creu i Sant Pau, Barcelona, España, 5Servicio de Psiquiatría. Hospital de la Santa Creu i Sant Pau, Barcelona, España, 6Servicio de Farmacia. Hospital de la Santa Creu i Sant Pau, Barcelona, España y 7Hospital de la Santa Creu i Sant Pau, Barcelona, España.
- 4008-3. Impacto de variantes sinónimas en las miocardiopatías hereditarias
- Ana Isabel Fernández Ávila1, Silvia Vilches Soria2, Irene Méndez Fernández2, Cristina Gómez González2, Renée Olsen Rodríguez3, Nélida Vázquez Aguilera2, Reyes Álvarez-García Revés4, Miriam Centeno Jiménez4, Constancio Medrano López4, Javier Bermejo Thomas2 y M.M. Ángeles Espinosa Castro2
1Cardiología. Centro de Investigación Biomédica en Red de Enfermedades Cardiovasculares (CIBER-CV), Madrid, España, 2Cardiología. Hospital General Universitario Gregorio Marañón, Madrid, España, 3Cardiología. Hospital Universitario de Getafe, Getafe (Madrid), España y 4Cardiología Pediátrica. Hospital General Universitario Gregorio Marañón, Madrid, España.
- 4008-4. Predictores de eventos adversos en patología aórtica. No solo el tamaño importa
- Ismael Arco Adamuz1, Miguel Morales García1, Inés Uribe Morales1, Diego Segura Rodríguez2, Eduardo Moreno Escobar2 y Rocío García Orta1
1Hospital Universitario Virgen de las Nieves, Granada, España y 2Hospital Clínico San Cecilio, Granada, España.
- 4008-5. Diferencias de género en el pronóstico aórtico en pacientes con síndrome de Marfan. Estudio longitudinal retrospectivo del registro REPAG
- Gisela Teixido Tura1, Diana Domingo Valero2, Rocío García Orta3, Francisco Calvo Iglesias4, Francisco Valera Martínez2, José María Larrañaga Moreira5, Daniel Martínez López6, Clara Badia Molins1, Rosario Sánchez Martínez7, Anna Sabaté Rotés8, Julián Palomino-Doza9, Javier Limeres Freire1, Eduardo Moreno Escobar3, Alberto Forteza Gil6 y Fernando Cabrera Bueno10
1Hospital Universitari Vall d'Hebron, Centro de Investigación Biomédica en Red, Enfermedades Cardiovasculares (CIBERCV), Barcelona, España, 2Hospital Universitario La Fe, Valencia, España, 3Hospital Universitario Virgen de las Nieves, Granada, España, 4Hospital Álvaro Cunqueiro, Vigo (Pontevedra), España, 5Complexo Hospitalario Universitario A Coruña, A Coruña, España, 6Hospital Universitario Puerta de Hierro, Madrid, España, 7Hospital General Universitario de Alicante, Alicante, España, 8Hospital Universitari Vall d'Hebron, Barcelona, España, 9Hospital Universitario 12 de Octubre, Madrid, España y 10Hospital Clínico Universitario Virgen de la Victoria, Málaga, España.
- 4008-6. ¿Es útil el screening familiar en miocardiopatía hipertrófica?
- Cristina Gómez González1, Alexis Rojas1, Silvia Vilches Soria1, Ana Paula Carrillo2, Teresa Bañuelos2, Anaëlle Garoux2, Irene Méndez Fernández1, Reyes Álvarez García-Rovés3, Miriam Centeno Jiménez3, Constancio Medrano López3, Ana Isabel Fernández Ávila4, Renée Olsen Rodríguez5, Nélida Vázquez Aguilera1, Javier Bermejo Thomas1 y M.M. Ángeles Espinosa Castro1
1Cardiología. Hospital General Universitario Gregorio Marañón, Madrid, España, 2Facultad de Medicina. Universidad Complutense, Madrid, España, 3Cardiología Pediátrica. Hospital General Universitario Gregorio Marañón, Madrid, España, 4CIBERCV. Hospital General Universitario Gregorio Marañón, Centro de Investigación Biomédica en Red, Enfermedades Cardiovasculares (CIBERCV), Madrid, España y 5Cardiología. Hospital Universitario de Getafe, Getafe (Madrid), España.
- 4008-7. Cohorte nacional de sospecha de miocarditis Pre-MYO: manejo asistencial en los 100 primeros casos
- Fernando Domínguez Rodríguez1, Alejandro Riquelme Pérez2, Ángela Carrillo Molina2, José María Larrañaga Moreira3, Jara Gayán Ordás4, Marcelo Sanmartín Fernández5, Belén Álvarez Álvarez6, Javier Torres Llergo7, Héctor Bueno Zamora8, Francisco Ridocci Soriano9, Elena Sufrate Sorzano10, Manuel Barreiro Pérez11, José María Viéitez Flórez12, Beatriz Fernández González13 y Domingo Pascual-Figal14
1Cardiología. Hospital Universitario Puerta de Hierro, Majadahonda (Madrid), España, 2Cardiología. Instituto Murciano de Investigación Biosanitaria Virgen de la Arrixaca, Murcia, España, 3Cardiología. Complexo Hospitalario Universitario A Coruña, A Coruña, España, 4Cardiología. Hospital Universitari Arnau de Vilanova, Lleida, España, 5Cardiología. Hospital Universitario Ramón y Cajal, Madrid, España, 6Cardiología. Complexo Hospitalario Universitario de Santiago de Compostela, Santiago de Compostela (A Coruña), España, 7Cardiología. Hospital Universitario de Jaén, Jaén, España, 8Cardiología. Hospital Universitario 12 de Octubre, Madrid, España, 9Cardiología. Hospital General Universitario, Valencia, España, 10Cardiología. Complejo Hospitalario San Millán-San Pedro, Logroño (La Rioja), España, 11Cardiología. Hospital Álvaro Cunqueiro, Vigo (Pontevedra), España, 12Cardiología. Hospital Universitario Lucus Augusti, Lugo, España, 13Cardiología. Hospital Universitario de Burgos, Burgos, España y 14Cardiología. Hospital Clínico Universitario Virgen de la Arrixaca, Murcia, España.
Más comunicaciones de los autores
- Álvarez-García Revés, Reyes
-
Bermejo Thomas, Javier
- 6030-178 - Predictores de recuperación de la función ventricular izquierda tras el implante de prótesis valvular aórtica percutánea
- 5018-2 - La esclerosis aórtica "funcional" se asocia a un aumento de riesgo de insuficiencia cardiaca y mortalidad
- 5018-5 - Efecto del bloqueo completo de rama izquierda como factor determinante de pronóstico en pacientes intervenidos de reemplazo valvular aórtico transcatéter. Experiencia de un centro terciario
- 5020-6 - Implante de válvula aórtica transcatéter en paciente joven: experiencia y factores predictores de eventos en un hospital terciario
- 6031-195 - Datos en la vida real de cangrelor en procedimientos de cardiología intervencionista
- 4016-7 - Gemelos digitales: nuevas estrategias personalizadas de ablación de fibrilación auricular
- 4008-6 - ¿Es útil el screening familiar en miocardiopatía hipertrófica?
- 5010-2 - ¿Refleja el ensayo DanGer shock la vida real?
- 5009-6 - Disfunción endotelial microvascular y espasmo microvascular en pacientes con infarto agudo de miocardio con elevación del ST y enfermedad multivaso: prevalencia y predictores clínicos y angiográficos
- 6110-9 - Estimación no invasiva de la presión capilar pulmonar mediante algoritmos de machine learning
- 5005-6 - Imagen de estasis mediante ecocardiografía para predecir el riesgo de ictus e infarto cerebral silente en los pacientes con miocardiopatía dilatada no isquémica
- 6055-364 - El gradiente de presión intraventricular es el índice ecocardiográfico más sensible del estado inotrópico del ventrículo izquierdo
- 6114-9 - Perfil clínico e impacto pronóstico de la anemia en los pacientes sometidos a implante de válvula aórtica transcatéter
- 4008-3 - Impacto de variantes sinónimas en las miocardiopatías hereditarias
- 6123-16 - Malrotación de dispositivos de asistencia ventricular izquierda tipo Impella
- 6105-3 - Canulación bilateral frente a unilateral en oxigenación con membrana extracorpórea veno-arterial periférica
- 5004-6 - Caracterización electrofisiológica del sustrato auricular mediante imagen electrocardiográfica (ECGi) sin necesidad de imagen cardiaca previa
- 5018-8 - Predictores de descompensación de insuficiencia cardiaca tras el implante de prótesis valvular aórtica percutánea
- Centeno Jiménez, Miriam
-
Espinosa Castro, M. Ángeles
- 4008-6 - ¿Es útil el screening familiar en miocardiopatía hipertrófica?
- 5002-11 - Caracterización y pronóstico de la amiloidosis cardiaca hereditaria por transtirretina en España
- 4008-3 - Impacto de variantes sinónimas en las miocardiopatías hereditarias
- 5005-6 - Imagen de estasis mediante ecocardiografía para predecir el riesgo de ictus e infarto cerebral silente en los pacientes con miocardiopatía dilatada no isquémica
- 5002-7 - Genotipos de alto riesgo en miocardiopatía dilatada: más allá de las arritmias
-
Fernández Ávila, Ana Isabel
- 5005-6 - Imagen de estasis mediante ecocardiografía para predecir el riesgo de ictus e infarto cerebral silente en los pacientes con miocardiopatía dilatada no isquémica
- 4008-3 - Impacto de variantes sinónimas en las miocardiopatías hereditarias
- 4008-6 - ¿Es útil el screening familiar en miocardiopatía hipertrófica?
- Gómez González, Cristina
- Medrano López, Constancio
- Méndez Fernández, Irene
-
Olsen Rodríguez, Renée
- 6048-306 - Perfil clínico y pronóstico de pacientes con infarto agudo de miocardio con arterias coronarias no obstruidas que hayan tenido un síndrome coronario agudo previo
- 6048-309 - El impacto de la enfermedad renal en pacientes con infarto agudo de miocardio con y sin lesiones obstructivas
- 6101-4 - Concordancia entre intervenciones cardiológicas y propuestas de inteligencia artificial para pacientes que presentan dolor torácico, basadas en interconsultas desde el servicio de urgencias a cardiología
- 6065-417 - Diferencias en cuanto al sexo en pacientes ingresados por insuficiencia cardiaca: características y pronóstico
- 4009-4 - Implicaciones diagnósticas y pronosticas de los niveles de biomarcadores de inflamación y disfunción endotelial en el infarto de miocardio con y sin arterias coronarias obstructivas
- 6042-279 - Toma de decisiones en los pacientes con dolor torácico y curva plana de troponina
- 4008-3 - Impacto de variantes sinónimas en las miocardiopatías hereditarias
- 6048-308 - Implicaciones pronósticas de los pacientes diabéticos con MINOCA
- 6060-376 - Resonancia magnética cardiaca en pacientes con MINOCA en la práctica clínica habitual
- 5019-5 - Factores predictores de MINOCA en pacientes con infarto agudo de miocardio
- 6013-83 - Características clínicas y pronóstico del MINOCA en función de la edad
- 6045-295 - Infarto agudo de miocardio ¿ha cambiado el manejo en los últimos años?
- 6072-450 - Strain global longitudinal en insuficiencia cardiaca con fracción de eyección preservada. ¿Es útil?
- 6076-474 - Factores predictivos de ingreso hospitalario en pacientes con dolor torácico y curva plana de troponina
- 6123-8 - Evolución a largo plazo y factores predictores en los pacientes con MINOCA
- 6063-404 - Impacto de la edad en la insuficiencia cardiaca. Particularidades clínicas, epidemiológicas y pronósticas en el paciente anciano
- 6010-46 - Diferencias entre hombres y mujeres menores de 65 años con infarto agudo de miocardio: una cohorte española
- 6006-19 - Evaluación pronóstica de un grupo de pacientes con dolor torácico y curva plana de troponina
- 6103-11 - MINOCA vs MICAD en el paciente anciano: características y pronóstico
- 6104-3 - Implementación de una herramienta de inteligencia artificial en una consulta de cardiología general. Una nueva oportunidad
- 6048-307 - Infarto de miocardio con arterias coronarias no obstructivas (MINOCA): características clínicas y pronóstico
- 4008-6 - ¿Es útil el screening familiar en miocardiopatía hipertrófica?
- 6063-406 - Impacto de la hiponatremia en la hospitalización por insuficiencia cardiaca
- Vázquez Aguilera, Nélida
- Vilches Soria, Silvia
