SEC 2021 - El Congreso de la Salud Cardiovascular
Introducción
Dr. Héctor Bueno
Presidente del Comité Científico del Congreso
Comité ejecutivo
Comité de evaluadores
Listado de sesiones
Índice de autores
5008. Cardiopatías familiares y genética cardiovascular I
5008-7. ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
Hospital Universitario Central de Asturias, Oviedo, Asturias.
Introducción y objetivos: La miocardiopatía hipertrófica (MCH) es la enfermedad cardiaca hereditaria más frecuente. A pesar de su definición como hereditaria con un patrón monogénico, en un 40-60% de los casos no se identifican variantes patogénicas en su exoma. Además hay estudios que apuntan a que hay otros factores genéticos que podrían explicar parte del riesgo de desarrollar MCH. Un grupo de genes candidatos a valorar son los que codifican long-non-coding RNA (lncRNA), que no se traducen a proteína y por tanto su variación escapa a las herramientas de predicción informática aplicables al exoma. Por lo tanto, el objetivo de este estudio sería evaluar la asociación de los lncRNA con el riesgo de desarrollar MCH.
Métodos: Secuenciamos una cohorte de 238 casos índice de MCH (58 ± 16 años; 63% varones) y 212 controles (70 ± 7 años; 45% varones) mediante un panel de 10 genes codificadores de lncRNA asociados a patología cardiovascular (H19, KCNQ1OT1, MHRT, CARMEN, FENDRR, TINCR, ANRIL, MIAT, PVT1, MALAT1), utilizando la tecnología de chips semiconductores en un equipo Ion GeneStudio S5 Sequencer (Ion Torrent).
Resultados: Una vez secuenciados, nos centramos en el lncRNA H19 como candidato para la identificación de polimorfismos de asociación con la MCH. Comparamos las frecuencias alélicas entre ambos grupos mediante un test Χ2 y solamente hemos encontrado diferencias significativas en el polimorfismo rs2075744 A/G (0,27 pacientes vs 0,40 controles; p < 0,0001; OR: 0,5665: IC: 0,4279-0,7500) (tabla).
|
Frecuencias del alelo minoritario, en pacientes y controles de los polimorfismos identificados en el gen H19 con una frecuencia mayor de 0,2 en nuestra cohorte (no había diferencias significativas en los menores de dicha frecuencia) |
|||
|
Polimorfismos |
MAF pacientes |
MAF controles |
p |
|
rs10840159 A/G |
0,44 |
0,47 |
0,3 |
|
rs2067051 C/T |
0,44 |
0,47 |
0,5 |
|
rs2075744 A/G |
0,27 |
0,40 |
0,0001 |
|
rs2075745 A/T |
0,44 |
0,47 |
0,4 |
|
rs2735469 G/A |
0,82 |
0,81 |
0,9 |
|
rs2839698 G/A |
0,47 |
0,49 |
0,6 |
|
rs2839701 C/G |
0,44 |
0,46 |
0,5 |
|
rs2839702 A/C |
0,44 |
0,47 |
0,4 |
|
rs2839703 T/C |
0,36 |
0,39 |
0,3 |
|
rs2839704 T/C |
0,36 |
0,39 |
0,3 |
|
rs3024270 C/G |
0,49 |
0,51 |
0,7 |
|
rs3741219 A/G |
0,45 |
0,48 |
0,4 |
|
MAF: Minor Allele Frequency. |
|||
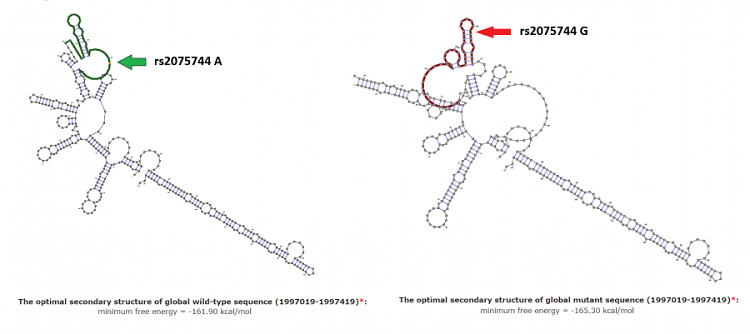
Diferencias estructurales en el lncRNA H19 en relación al polimorfismo rs2075744 A/G. El plegamiento estructural del H19 se predijo con http://rth.dk/resources/rnasnp/. Las flechas indican el cambio de nucleótido A/G.
Conclusiones: En conclusión, se ha observado que la frecuencia del alelo minoritario (G) en el polimorfismo rs2075744 A/G es mucho menor en pacientes que en controles. De esta forma, hace pensar que este alelo podría disminuir el riesgo de desarrollar MCH, y jugar un papel protector frente a dicha patología. Esta variante podría suponer un cambio en la estructura del H19 que modificaría la regulación de la expresión génica controlada por este lncRNA (fig.). Sin embargo, la relevancia funcional de este cambio requiere validación experimental.
Comunicaciones disponibles de "Cardiopatías familiares y genética cardiovascular I"
- 5008-1. MODERADORA
- Rosa Macías Ruiz, Granada
- 5008-2. LAS VÍAS WNT, HIPPO Y ADIPOGÉNESIS, TAMBIÉN ALTERADAS EN GRASA EPICÁRDICA DE PACIENTES CON MIOCARDIOPATÍA ARRITMOGÉNICA A TRAVÉS DE LA DISREGULACIÓN DE MIRNA
- Aitana Braza-Boils1, Julia Martínez-Solé2, Pilar Molina3, Juan Giner3, Izaskun Morillo4, Elena Martos-Albero4, Yolanda Abellán Pinar3, Jennifer Sancho3, Cristian Soto-Moh5, Jorge Sanz-Sánchez6, Luis Martínez-Dolz7 y Esther Zorio8
1Grupo Acreditado CaFaMuSME. Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, CIBERCV, ISCIII Valencia. 2Servicio de Cardiología, Hospital Universitario La Fe, Valencia. 3Instituto de Medicina Legal y Forense, Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Valencia. 4Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Valencia. 5Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Instituto de Medicina Legal y Forense, Valencia. 6Servicio de Cardiología, Hospital Universitario La Fe, Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Valencia. 7Servicio de Cardiología, Hospital Universitario La Fe, CIBERCV, ISCIII, Valencia. 8Servicio de Cardiología, Hospital Universitario La Fe, Grupo CaFaMuSME. Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, CIBERCV, ISCIII, Valencia.
- 5008-3. MECANISMOS ARRITMOGÉNICOS EN UN MODELO ANIMAL DEL SÍNDROME DE QT CORTO TIPO 3
- Ana Isabel Moreno Manuel1, Álvaro Macías Martínez1, Francisco Miguel Cruz Uréndez1, Lilian Karina Gutiérrez Espinosa de los Monteros1, Isabel Martínez Carrascoso1, Francisco José Bermúdez Jiménez2, María Linarejos Vera Pedrosa1, Juan Antonio Bernal1 y José Jalife1
1Centro Nacional de Investigaciones Cardiovasculares, Madrid. 2Hospital Universitario Virgen de las Nieves, Granada.
- 5008-4. EN BUSCA DE TEJIDOS SUBROGADOS PARA EL MIOCARDIO: LA METILACIÓN DEL PROMOTOR DEL GEN DE DESMOPLAQUINA EN QUERATINOCITOS BUCALES CORRELACIONA CON LA DEL MIOCARDIO
- Aitana Braza-Boils1, Liliane Lin Wong2, Tomás Luján3, Luis Almenar Bonet4, Raquel López-Vilella4, Ignacio Sánchez-Lázaro4, Víctor Donoso5, Julia Martínez-Solé5, Tomás Heredia6, Lucía Doñate Bertolín6, Luis Martínez-Dolz7 y Esther Zorio8
1Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, CIBERCV, ISCIII, Valencia. 2Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Valencia. 3Grupo Acreditado CaFaMuSME, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Servicio de Cardiología, Hospital Universitario La Fe, Valencia. 4Unidad de Insuficiencia Cardiaca y Trasplante, Servicio de Cardiología, Hospital Universitario La Fe, Valencia. 5Servicio de Cardiología, Hospital Universitario La Fe, Valencia. 6Servicio de Cirugía Cardiovascular, Hospital Universitario La Fe, Valencia. 7Servicio de Cardiología, Hospital Universitario La Fe, CIBERCV, ISCIII, Valencia. 8Servicio de Cardiología, Hospital Universitario La Fe, Grupo CaFaMuSME, CIBERCV, ISCIII, Fundación para la Investigación del Hospital Universitario y Politécnico La Fe, Valencia.
- 5008-5. LA VARIANTE ALÉLICA SMAD2RS29725537:C> A SE ASOCIA CON EL ORIGEN ANÓMALO ELEVADO DE LAS ARTERIAS CORONARIAS EN EL RATÓN DE LABORATORIO
- María Carmen Fernández, Miguel A. López-Unzu López, María Teresa Soto-Navarrete, Laura Martín-Chaves, Bárbara Pozo-Vilumbrales, Ana Carmen Durán y Borja Fernández
Universidad de Málaga.
- 5008-6. MUTACIONES MISSENSE DEL DOMINIO ROD2 DEL GEN FLNC SE ASOCIAN A UN FENOTIPO CARDIACO CARACTERÍSTICO EN «DIENTES DE TIBURÓN» CON UN REMODELADO CARDIACO DISTINTIVO
- Francisco José Bermúdez Jiménez1, Víctor Carriel2, Adrián Fernández3, Roberto Barriales-Villa4, Soledad García Hernández5, Francisco Miguel Cruz6, Andreas Brodehl7, Rosa Macías-Ruiz1 y Juan Jiménez Jáimez1
1Hospital Universitario Virgen de las Nieves, Granada. 2Universidad de Granada. 3Fundación Favaloro, Buenos Aires (Argentina). 4Complexo Hospitalario Universitario A Coruña. 5Health in Code, A Coruña. 6Centro Nacional de Investigaciones Cardiovasculares, Madrid. 7Bielefeld University, Bad Oeynhausen, Nordrhein-Westfalen (Alemania).
- 5008-7. ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
- Elías Cuesta Llavona, Rebeca Lorca Gutiérrez, Israel David Duarte Herrera, Beatriz Díaz-Molina, José Luis Lambert Rodríguez, Sara Iglesias Álvarez, Belén Alonso González, José Julián Rodríguez Reguero, Eliecer Coto García y Juan Gómez de Oña
Hospital Universitario Central de Asturias, Oviedo, Asturias.
- 5008-8. CARACTERIZACIÓN CLÍNICA DE LA AMILOIDOSIS CARDIACA EN UNA COHORTE CONTEMPORÁNEA: RESULTADOS DEL REGISTRO PROSPECTIVO MULTICÉNTRICO AMI-GAL (REGISTRO DE AMILOIDOSIS CARDIACA EN GALICIA)
- Gonzalo Barge Caballero1, Eduardo Barge Caballero1, Manuel López Pérez2, Raquel Bilbao Quesada3, Eva González Babarro4, Inés Gómez Otero5, Andrea López López6, Mario Gutiérrez Feijoo7, Alfonso Varela Román5, Carlos González Juanatey6, Óscar Díaz Castro3 y María G. Crespo Leiro1
1Complexo Hospitalario Universitario A Coruña. 2Hospital Arquitecto Marcide, Ferrol, A Coruña. 3Hospital Universitario Alvaro Cunqueiro, Vigo, Pontevedra. 4Complejo Hospitalario de Pontevedra. 5Complexo Hospitalario Universitario de Santiago de Compostela, A Coruña. 6Hospital Universitario Lucus Augusti, Lugo. 7Complexo Hospitalario de Ourense.
Más comunicaciones de los autores
- Alonso González, Belén
- Coto García, Eliecer
-
Cuesta Llavona, Elías
- 5015-4 - RELACIÓN DE LA MICROBIOTA INTESTINAL Y LA ACTIVIDAD NEUROHORMONAL E INFLAMATORIA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
- 6039-9 - IMPACTO DE LA MICROBIOTA INTESTINAL EN EL PRONÓSTICO DE LA INSUFICIENCIA CARDIACA
-
Díaz Molina, Beatriz
- 6026-8 - SEGURIDAD DE LAS VACUNAS CONTRA LA COVID-19 EN TRASPLANTADOS CARDIACOS
- 6006-6 - CONCORDANCIA INTEROBSERVADORES EN LA REVISIÓN DE HISTORIAS CLÍNICAS PREVIA A LA RECOGIDA SISTEMÁTICA DE DATOS PARA EL ESTUDIO DE LA VALIDEZ DEL CONJUNTO MÍNIMO BÁSICO DE DATOS COMO FUENTE PARA LA INVESTIGACIÓN DE RESULTADOS EN LA ATENCIÓN A LA INSUFICIENCIA CARDIACA
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
- 4025-7 - ESTUDIO MULTICÉNTRICO ESPAÑOL SOBRE LA MONITORIZACIÓN E INCIDENCIA DE HIPERPOTASEMIA EN PACIENTES CON INSUFICIENCIA CARDIACA Y FRACCIÓN EYECCIÓN REDUCIDA. SPANIK-HF
- 4025-6 - SPANIK-HF-ESTUDIO MULTICÉNTRICO ESPAÑOL SOBRE LA PREVALENCIA, INCIDENCIA Y PRONÓSTICO DE HIPERPOTASEMIA EN PACIENTES CON INSUFICIENCIA CARDIACA: DATOS BASALES
- 4025-4 - INCIDENCIA Y PRONÓSTICO DEL CÁNCER COLORRECTAL TRAS EL TRASPLANTE CARDIACO. ESTUDIO MULTICÉNTRICO ESPAÑOL
- Duarte Herrera, Israel David
-
Gómez de Oña, Juan
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
- 5015-4 - RELACIÓN DE LA MICROBIOTA INTESTINAL Y LA ACTIVIDAD NEUROHORMONAL E INFLAMATORIA EN PACIENTES CON INSUFICIENCIA CARDIACA
- 6039-9 - IMPACTO DE LA MICROBIOTA INTESTINAL EN EL PRONÓSTICO DE LA INSUFICIENCIA CARDIACA
- Iglesias Álvarez, Sara
-
Lambert Rodríguez, José Luis
- 5004-3 - INFECCIÓN POR SARS-COV-2 EN PACIENTES CON TRASPLANTE CARDIACO EN ESPAÑA. EPIDEMIOLOGÍA, MANEJO Y PRONÓSTICO. ESTUDIO MULTICÉNTRICO
- 6026-8 - SEGURIDAD DE LAS VACUNAS CONTRA LA COVID-19 EN TRASPLANTADOS CARDIACOS
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
-
Lorca Gutiérrez, Rebeca
- 6040-14 - EVOLUCIÓN A LARGO PLAZO DE PACIENTES CON MIOCARDIOPATÍA HIPERTRÓFICA PORTADORES DE DESFIBRILADOR AUTOMÁTICO IMPLANTABLE EN PREVENCIÓN PRIMARIA Y SECUNDARIA
- 5029-2 - HISTORIA NATURAL DE LA MIOCARDIOPATÍA DILATADA POR VARIANTES EN MYH7
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
-
Rodríguez Reguero, José Julián
- 6040-14 - EVOLUCIÓN A LARGO PLAZO DE PACIENTES CON MIOCARDIOPATÍA HIPERTRÓFICA PORTADORES DE DESFIBRILADOR AUTOMÁTICO IMPLANTABLE EN PREVENCIÓN PRIMARIA Y SECUNDARIA
- 5008-7 - ASOCIACIÓN DEL POLIMORFISMO RS2075744 EN EL ARN LARGO NO CODIFICANTE H19 CON EL RIESGO DE DESARROLLAR MIOCARDIOPATÍA HIPERTRÓFICA
