SEC 2013 - El Congreso de las Enfermedades Cardiovasculares
4021. Arritmias ventriculares. Ablación y técnicas de imagen
4021-6. Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
Introducción: Las secuencias de resonancia magnética (RMN) con realce tardío adquiridas durante apnea (RMN-2D) representan el estándar en la caracterización del tejido miocárdico. Sin embargo, estas secuencias presentan problemas para la reconstrucción del volumen 3D debido al desplazamiento entre los diferentes cortes y su limitada resolución en el eje Z. Esto limita su utilización en la identificación de canales de conducción (CC), objetivo de la ablación de taquicardia ventricular (TV). Este trabajo tiene como objetivo determinar si la utilización de una secuencia con navegador (RMN-3D) permite identificar mejor los CC debido a la ausencia de desplazamiento entre imágenes y su mayor resolución espacial.
Métodos: Se incluyeron 30 pacientes con cardiopatía estructural a los que se les practicó un estudio de RMN previo a la ablación de TV. La resolución espacial de la RMN-2D fue de 1,4 × 1,4 × 7 mm mientras que la de la secuencia RMN-3D fue de 1,4 × 1,4 × 1,4 mm. La masa de cicatriz (diferenciada entre cicatriz densa y zona periinfarto) fue medida. Se realizó la reconstrucción 3D del ventrículo izquierdo en las dos secuencias de RMN, se identificaron los CC y se compararon con los CC identificados en los mapas electroanatómicos.
Resultados: No se observaron diferencias entre la masa de cicatriz del tejido necrótico entre las secuencias RMN-2D y RMN-3D (7,48 ± 6,68 vs 6,26 ± 4,37 gramos, respectivamente; p = 0,264). Sin embargo, se observó una mayor cuantificación de masa de zona periinfarto en la secuencia RMN-3D respecto la secuencia RMN-2D (10,92 ± 5,98 vs 9,22 ± 5,97 gramos, respectivamente; p = 0,028). En la secuencia RMN-3D se identificó un número de CC similar al total de CC observados en los mapas electroanatómicos (2,47 ± 1,25 vs 2,55 ± 1,64; p = 0,650), mientras que en la secuencia RMN-2D el número de CC observados fue menor (2,08 ± 1,26 vs 2,55 ± 1,64; p = 0,050).
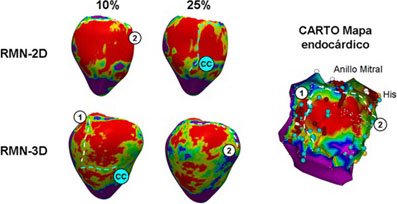
Conclusiones: La mayor resolución espacial de la secuencia RMN-3D permiten diferenciar un mayor número de CC que la secuencia RMN-2D.
Comunicaciones disponibles de "Arritmias ventriculares. Ablación y técnicas de imagen"
- 4021-1. Presentación
- Ernesto Díaz Infante, Sevilla e Ignacio Fernández Lozano, Madrid.
- 4021-2. Ablación de sustrato endoepicárdica en la miocardiopatía arritmogénica. Estudio prospectivo multicéntrico
- Juan Fernández-Armenta1, Lluis Mont2, Luis Tercedor3, Alonso Pedrote4, Ignasi Anguera1, Alberto Barrera1 y Antonio Berruezo1 del 1Servicio de Cardiología, Unidad de Arritmias, Hospital Clínic de Barcelona, 2 Servicio de Cardiología, Unidad de Arritmias, Hospital Clínic de Barcelona, 3Hospital Universitario Virgen de las Nieves, Granada y 4Hospital Universitario Virgen del Rocío, Sevilla.
- 4021-3. Eficacia y seguridad de la ablación epicárdica de arritmias ventriculares
- Ana Cecilia Gonzales Luna, Javier Moreno Planas, Lázaro Hernández Jiménez, Yolanda Abreu Hernández, Laila González Melchor, Victoria Cañadas Godoy, Nicasio Pérez Castellano y Julián Pérez Villacastín del Hospital Clínico San Carlos, Madrid.
- 4021-4. ¿Procainamida IV vs Amiodarona IV en el tratamiento agudo de las taquicardias de QRS ancho? Evidencia necesaria para recomendaciones efectivas (Estudio PROCAMIO)
- Mercedes Ortiz Patón, Jesús Almendral Garrote, Alfonso Martín Martínez, Rafael Peinado Peinado, Fernando Arribas Ynsaurriaga, Blanca Coll-Vinent y Carmen del Arco del Grupo PROCAMIO.
- 4021-5. La ablación por radiofrecuencia de extrasistolia ventricular frecuente puede evitar implantes innecesarios de DAI
- Andrés Díaz Garzón1, Diego Penela1, Luis Aguinaga2, Juan Fernández-Armenta1, Lluís Mont1, Josep Brugada1, Katja Zeppenfeld3 y Antonio Berruezo1 del 1Sección de Arritmias, Servicio de Cardiología, ICT, Hospital Clínic, Universitat de Barcelona, 2Hospital Argerich, Buenos Aires, Argentina, Ciudad Autónoma de Buenos Aires (Buenos Aires) y 3Department of Cardiology, Leiden University Medical Center, Leiden (Netherlands Antilles (General)).
- 4021-6. Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- David Andreu Caballero1, José Tomás Ortiz Pérez2, Rosario Jesús Perea Palazón3, Teresa María de Caralt Robira3, Juan Fernández-Armenta Pastor1, Josep Lluís Mont Girbau1, Josep Brugada Terradellas1 y Antonio Berruezo Sánchez1 de la 1Sección de Arritmias, Servicio de Cardiología, 2Servicio de Cardiología y 3Centro Diagnóstico de la Imagen, Hospital Clínic, Universitat de Barcelona.
- 4021-7. Análisis de episodios de fibrilación auricular paroxística detectados mediante monitorización remota con holter subcutáneo en pacientes con ictus isquémico criptogénico
- María Gracia Sandín Fuentes1, Esther Rojo-Martínez2, Emilio García Morán1, Jerónimo Rubio Sanz1, M. Ruiz Pinero2, Juan Manuel Villadeamigo Romero1, J.F. Arenillas Lara2 y J. Alberto San Román Calvar1 del 1Instituto de Ciencias del Corazón (ICICOR), Valladolid y 2Servicio de Neurología, Hospital Clínico Universitario, Valladolid.
Más comunicaciones de los autores
-
Andreu Caballero, David
- 6000-51 - Resultados a largo plazo de la ablación de TV en pacientes con cardiopatía estructural y fracción de eyección ¿ 35%. ¿Podría la ablación ser una alternativa al implante de DAI?
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
-
Berruezo Sánchez, Antonio
- 6000-51 - Resultados a largo plazo de la ablación de TV en pacientes con cardiopatía estructural y fracción de eyección ¿ 35%. ¿Podría la ablación ser una alternativa al implante de DAI?
- 4022-3 - ReAblación de fibrilación auricular guiada por resonancia magnética: Localización y ablación anatómica de Gaps
- 6000-52 - Caracterización de las lesiones y gaps tras crioablación de fibrilación auricular mediante resonancia magnética
- 6000-50 - Inhibidor tisular plasmático de metaloproteinasas de matriz-1 (TIMP-1). Un poderoso predictor de mortalidad a largo plazo en pacientes tratados con terapia de resincronización cardiaca
- 6000-47 - Ablación del nodo AV para optimizar la terapia de desincronización en pacientes con miocardiopatía hipertrófica obstructiva
- 6000-65 - Caracterización de las lesiones de crioablación vs radiofrecuencia mediante resonancia magnética en pacientes sometidos a ablación de fibrilación auricular
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- 4021-5 - La ablación por radiofrecuencia de extrasistolia ventricular frecuente puede evitar implantes innecesarios de DAI
- 6000-35 - Distribución por sexos del tamaño de la cicatriz miocárdica por resonancia magnética en pacientes sometidos a terapia de resincronización cardiaca (TRC) y su relación con el riesgo arrítmico
- 4021-2 - Ablación de sustrato endoepicárdica en la miocardiopatía arritmogénica. Estudio prospectivo multicéntrico
-
Brugada Terradellas, Josep
- 6018-582 - Pronóstico de la insuficiencia cardiaca de diagnóstico ambulatorio en nuestro medio: la fracción de eyección preservada tiene un pronóstico cardiovascular comparable a la reducida
- 4021-5 - La ablación por radiofrecuencia de extrasistolia ventricular frecuente puede evitar implantes innecesarios de DAI
- 6000-35 - Distribución por sexos del tamaño de la cicatriz miocárdica por resonancia magnética en pacientes sometidos a terapia de resincronización cardiaca (TRC) y su relación con el riesgo arrítmico
- 6000-47 - Ablación del nodo AV para optimizar la terapia de desincronización en pacientes con miocardiopatía hipertrófica obstructiva
- 6000-51 - Resultados a largo plazo de la ablación de TV en pacientes con cardiopatía estructural y fracción de eyección ¿ 35%. ¿Podría la ablación ser una alternativa al implante de DAI?
- 6000-50 - Inhibidor tisular plasmático de metaloproteinasas de matriz-1 (TIMP-1). Un poderoso predictor de mortalidad a largo plazo en pacientes tratados con terapia de resincronización cardiaca
- 4001-5 - Papel de la metaloproteinasa de matriz 2 como marcador pronóstico en pacientes que debutan ambulatoriamente de insuficiencia cardiaca
- 4002-5 - Utilidad de las metaloproteinasas de matriz para el diagnóstico ambulatorio de la insuficiencia cardiaca: correlación con parámetros ecocardiográficos y péptido natriurético tipo B
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
-
de Caralt Robira, Teresa María
- 4039-4 - Estudio aleatorizado de la evaluación del dolor torácico agudo mediante ecocardiografía de ejercicio o tomografía cardiaca multidetector
- 6000-65 - Caracterización de las lesiones de crioablación vs radiofrecuencia mediante resonancia magnética en pacientes sometidos a ablación de fibrilación auricular
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- 6000-52 - Caracterización de las lesiones y gaps tras crioablación de fibrilación auricular mediante resonancia magnética
- 4022-3 - ReAblación de fibrilación auricular guiada por resonancia magnética: Localización y ablación anatómica de Gaps
-
Fernández-Armenta Pastor, Juan
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- 6000-51 - Resultados a largo plazo de la ablación de TV en pacientes con cardiopatía estructural y fracción de eyección ¿ 35%. ¿Podría la ablación ser una alternativa al implante de DAI?
- 4021-5 - La ablación por radiofrecuencia de extrasistolia ventricular frecuente puede evitar implantes innecesarios de DAI
- 6000-35 - Distribución por sexos del tamaño de la cicatriz miocárdica por resonancia magnética en pacientes sometidos a terapia de resincronización cardiaca (TRC) y su relación con el riesgo arrítmico
- 4021-2 - Ablación de sustrato endoepicárdica en la miocardiopatía arritmogénica. Estudio prospectivo multicéntrico
-
Mont Girbau, Lluís
- 6000-51 - Resultados a largo plazo de la ablación de TV en pacientes con cardiopatía estructural y fracción de eyección ¿ 35%. ¿Podría la ablación ser una alternativa al implante de DAI?
- 6000-48 - La geometría auricular se asocia a historia de ictus previo en pacientes sometidos a ablación de fibrilación auricular
- 6000-35 - Distribución por sexos del tamaño de la cicatriz miocárdica por resonancia magnética en pacientes sometidos a terapia de resincronización cardiaca (TRC) y su relación con el riesgo arrítmico
- 6000-50 - Inhibidor tisular plasmático de metaloproteinasas de matriz-1 (TIMP-1). Un poderoso predictor de mortalidad a largo plazo en pacientes tratados con terapia de resincronización cardiaca
- 6000-47 - Ablación del nodo AV para optimizar la terapia de desincronización en pacientes con miocardiopatía hipertrófica obstructiva
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- 4021-5 - La ablación por radiofrecuencia de extrasistolia ventricular frecuente puede evitar implantes innecesarios de DAI
- 4022-3 - ReAblación de fibrilación auricular guiada por resonancia magnética: Localización y ablación anatómica de Gaps
- 6007-294 - Remodelado auricular reverso volumétrico y funcional tras la terapia de resincronización cardiaca: Relación con la respuesta clínica a la terapia
- 4021-2 - Ablación de sustrato endoepicárdica en la miocardiopatía arritmogénica. Estudio prospectivo multicéntrico
- 6000-65 - Caracterización de las lesiones de crioablación vs radiofrecuencia mediante resonancia magnética en pacientes sometidos a ablación de fibrilación auricular
- 6000-52 - Caracterización de las lesiones y gaps tras crioablación de fibrilación auricular mediante resonancia magnética
-
Ortiz Pérez, José Tomás
- 4021-6 - Las secuencias de resonancia 3D mejoran la identificación de los canales de conducción en la ablación de taquicardias ventriculares
- 4039-4 - Estudio aleatorizado de la evaluación del dolor torácico agudo mediante ecocardiografía de ejercicio o tomografía cardiaca multidetector
- 6003-201 - Beneficio de la hipotermia terapéutica independientemente del primer ritmo registrado en pacientes en coma posparo cardiaco
- Perea Palazón, Rosario Jesús
