SEC 2020 - El e-Congreso de la Salud Cardiovascular
Introducción
Dr. Héctor Bueno
Presidente del Comité Científico del Congreso
Comité ejecutivo
Comité de evaluadores
Listado de sesiones
Índice de autores
5016. Cardiopatías familiares, muerte súbita y genética cardiovascular
5016-6. ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
Complejo Hospitalario de Toledo.
Introducción y objetivos: La hipercolesterolemia familiar (HF) es una enfermedad genética autosómica dominante del metabolismo lipídico, caracterizada por niveles elevados de colesterol de lipoproteínas de baja densidad (LDLc) y elevado riesgo de enfermedad cardiovascular ateroesclerótica. Se produce típicamente por mutaciones en el gen del receptor de LDL (LDLR), aunque pueden observarse otras alteraciones. El objetivo de este trabajo es analizar los trastornos genéticos de HF hallados en el área sanitaria de un hospital de tercer nivel.
Métodos: Evaluamos las variantes en los genes LDLR, APOB, APOE, PCSK9, STAP1, LDLRAP1 y LIPA obtenidas en 84 individuos con diagnóstico clínico de HF en el área sanitaria de un hospital de tercer nivel, a partir de una muestra de 752 pacientes con perfil lipídico alterado obtenida mediante una estrategia de cribado a partir de analítica centralizada preexistente.
Resultados: Se obtuvo diagnóstico clínico en el 17,9% de los evaluados, con una tasa de positividad genética del 70,2%, que presentaban un LDL colesterol de 305,7 mg/dl IC95% (250,4; 360,9 mg/dl). Se identificaron 50 diferentes tipos de mutaciones, sobre todo en LDLR. Las variantes patogénicas más frecuentes fueron c.1342C > T y c.313+1G > C. Se objetivó que las variantes nulas tienen un perfil fenotípico más grave.
|
Medias y DE de perfil lipídico y puntuación DLCN en función de tipo de alelo, efecto-localización y clase patogénica |
||||||
|
Tipo de alelo |
N |
Col total (mg/dl) ± DE |
HDLC (mg/dl) ± DE |
Triglicéridos (mg/dl) ± DE |
LDLC (mg/dl) ± DE |
DLCN (ptos) ± DE |
|
Defectuoso |
19 |
339 (291,13-386,87) |
53,10 (42,98-63,22) |
110,89 (71,46-150,32) |
287,57 (244,18-330,96) |
7,31 (5,03-9,59) |
|
No disponible |
18 |
337,38 (302,12-372,64) |
53,22 (38,18-68,26) |
128,72 (90,41-167,03) |
305,61 (253,12-358,10) |
7,33 (6,10-8,56) |
|
Nulo |
22 |
355,68 (311,98-399,38) |
52,91 (36,16-69,66) |
126,81 (86,08-167,54) |
321,41 (257,83-384,99) |
8,22 (6,09-10,35) |
|
Efecto |
||||||
|
Exonic-Del |
3 |
400,0 (377,29-422,71) |
46,66 (42,05-51,27) |
147,33 (95,29-199,37) |
363,33 (288,79-437,87) |
8 (6,27-9,73) |
|
Exonic-Deletion-InFrame |
1 |
442 |
99 |
118 |
319 |
6 |
|
Exonic-Synonymous |
1 |
382 |
49 |
190 |
295 |
7 |
|
Intronic-Splicing |
9 |
347,11 (295,0-399,22) |
48,11 (41,21-55,01) |
112,0 (70,02-153,98) |
340,77 (292,29-389,25) |
9,55 (7,54-11,56) |
|
Missense |
32 |
334,62 (297,61-371,63) |
53,37 (42,45-64,29) |
117,15 (83,13-151,17) |
290,34 (240,79-339,89) |
7,03 (5,50-8,56) |
|
Nonsense |
12 |
345,83 (305,35-386,31) |
53,25 (32,17-74,33) |
126,0 (80,95-171,05) |
306,16 (244,10-368,22) |
8,08 (5,66-10,60) |
|
Promotor |
1 |
333 |
63 |
195 |
300 |
7 |
|
Clase patogénica |
||||||
|
Clase I |
32 |
351,09 (300,04-402,14) |
53,03 (37,05-69,01) |
117,75 (79,25-156,25) |
319,68 (260,66-378,70) |
8,25 (5,89-10,61) |
|
Clase II |
17 |
335,52 (307,49-363,55) |
53,88 (43,48-64,28) |
107,88 (73,82-141,94) |
289,64 (238,34-340,94) |
7,0 (5,89-8,11) |
|
Clase III |
10 |
340,0 (305,99-374,01) |
51,80 (37,25-66,35) |
161,20 (132,12-190,28) |
288,20 (249,48-236,92) |
6,90 (5,91-7,89) |
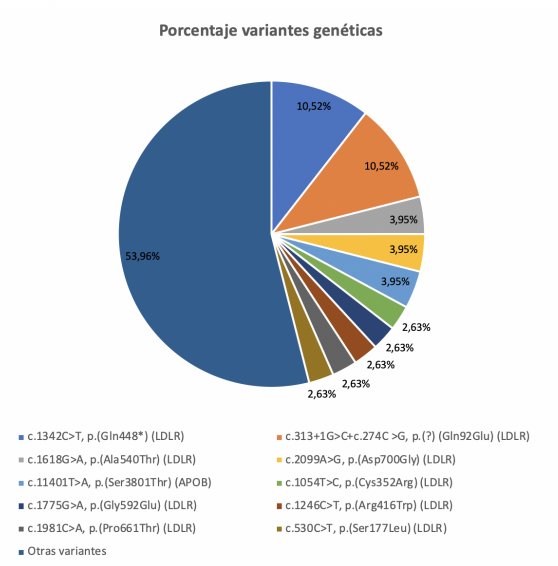
Porcentaje de variantes genéticas más frecuentes de la cohorte.
Conclusiones: Se ha detectado una elevada tasa de alteración genética. En la mayoría de los casos, las alteraciones fenotípicas no permiten predecir los hallazgos genéticos, que añaden información sobre el riesgo de los pacientes.
Comunicaciones disponibles de "Cardiopatías familiares, muerte súbita y genética cardiovascular"
- 5016-2. CRIBADO ACTIVO Y SECUENCIAL DE CASOS ÍNDICES DE HIPERCOLESTEROLEMIA FAMILIAR A PARTIR DE DATOS ANALÍTICOS CENTRALIZADOS
- Fernando Sabatel Pérez1, Joaquín Sánchez-Prieto Castillo1, Helena Contreras Mármol1, Víctor Manuel Becerra Muñoz2, Pedro Mata López3 y Luis Rodríguez Padial1
1Complejo Hospitalario de Toledo, SESCAM, Toledo. 2Hospital Clínico Universitario Virgen de la Victoria, Málaga. 3Fundación Hipercolesterolemia Familiar, Madrid.
- 5016-3. SUPERIORIDAD DE LOS CRITERIOS INMUNOHISTOQUÍMICOS SOBRE LOS CRITERIOS DE DALLAS EN EL DIAGNÓSTICO DE MIOCARDITIS Y MIOCARDIOPATÍA INFLAMATORIA
- Fernando Domínguez Rodríguez1, Rafael José Cobas Paz2, Juan Francisco Oteo Domínguez1, Francisco Hernández Pérez1, Manuel Gómez Bueno1, Clara Salas Antón1, Marta Cobo Marcos1, Esther González López1, Luis Enrique Escobar López1, Silvia Vilches Soria1, Cristina Daniela Mitroi1, Pablo García Pavía1 y Javier Segovia Cubero1
1Hospital Universitario Puerta de Hierro, Majadahonda (Madrid). 2Complexo Hospitalario Universitario de Vigo-Xeral-Cíes, Vigo (Pontevedra).
- 5016-4. EVENTOS EMBÓLICOS EN PACIENTES CON AMILOIDOSIS CARDIACA POR TRANSTIRETINA SIN FIBRILACIÓN AURICULAR
- Silvia Vilches Soria1, Esther González López1, Fernando Domínguez Rodríguez1, Luis Enrique Escobar López1, Adrián Rivas Pérez1, Aitor Hernández1, Stefano Perlini2, Claudio Rapezzi3, Mathew Maurer4, Julián Gillmore5 y Pablo García Pavía1
1Hospital Universitario Puerta de Hierro, Majadahonda (Madrid). 2Universidad de Pavía (Italia). 3Universidad de Bolonia (Italia). 4Hospital Universitario de Columbia, Nueva York. 5Centro Nacional de Amiloidosis, Londres (Reino Unido).
- 5016-5. PERFIL CLÍNICO Y PRONÓSTICO DE LA MIOCARDITIS Y MIOCARDIOPATÍA INFLAMATORIA CONFIRMADA POR BIOPSIA ENDOMIOCÁRDICA
- Fernando Domínguez Rodríguez1, Rafael José Cobas Paz2, Juan Francisco Oteo Domínguez1, Francisco José Hernández Pérez1, Manuel Gómez Bueno1, Clara Salas Antón1, Marta Cobo Marcos1, Esther González López1, Luis Enrique Escobar López1, Silvia Vilches Soria1, Cristina Daniela Mitroi1, Pablo García Pavía1 y Javier Segovia Cubero1
1Hospital Universitario Puerta de Hierro, Majadahonda (Madrid). 2Complexo Hospitalario Universitario de Vigo-Xeral-Cíes, Vigo (Pontevedra).
- 5016-6. ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- Joaquín Sánchez-Prieto Castillo, Fernando Sabatel Pérez, Helena Contreras Mármol, Carlos de Cabo Porras, Alejandro Gadella Fernández y Luis Rodríguez Padial
Complejo Hospitalario de Toledo.
- 5016-7. LA DISFUNCIÓN DE KIR2,1 SUBYACE A LOS DEFECTOS ELECTROMECÁNICOS QUE CONDUCEN A ARRITMIAS EN UN MODELO DE RATÓN DEL SÍNDROME DE ANDERSEN-TAWIL TIPO 1
- Álvaro Macías Martínez, Andrés González-Guerra, Ana Isabel Moreno Manuel, Francisco Miguel Cruz Uréndez, Francisco José Bermúdez Jiménez, Lilian Karina Gutiérrez-Espinosa de los Monteros, María Linarejos Vera-Pedrosa, Vicente Andrés, Juan A. Bernal y José Jalife
Centro Nacional de Investigaciones Cardiovasculares (CNIC), Madrid.
- 5016-8. PAPEL DEL ENTRECRUZAMIENTO DEL COLÁGENO EN LA AFECTACIÓN MIOCÁRDICA PRIMARIA EN EL SÍNDROME DE MARFAN: ESTUDIO DE BIOMARCADORES
- Eusebio García-Izquierdo Jaén1, Susana Mingo Santos1, Xabier Cia Mendioroz1, Vanessa Moñivas Palomero1, Carlos Esteban Martín López1, Sara Navarro Rico1, Silvia Rosado García2, María Gil Ligero2, Antonio J. Sánchez López2, Miguel A. Cavero Gibanel1, Juan Miguel Redondo3, Javier Segovia Cubero1 y Alberto Forteza Gil1
1Hospital Universitario Puerta de Hierro, Majadahonda (Madrid). 2Instituto de Investigación Sanitaria Puerta de Hierro-Segovia de Arana. 3Centro Nacional de Investigaciones Cardiovasculares (CNIC), Madrid.
- 5016-9. MIOCARDIOPATÍA ARRITMOGÉNICA CON AFECTACIÓN PREDOMINANTE DE VENTRÍCULO IZQUIERDO. CARACTERÍSTICAS CLÍNICAS Y PRONÓSTICO
- José Miguel Martín Torres1, Eloísa Feliu Rey2, Amaya García-Fernández1, Laura García-Cano1, Rafal Mosciki3, Luna Carrillo Alemán4, Juan Gabriel Martínez Martínez1 y Juan Miguel Ruiz Nodar1
1Servicio de Cardiología, Hospital General Universitario de Alicante. 2Unidad de Resonancia Magnética, INSCANNER, Alicante. 3Servicio de Cardiología, Hospital General de Elda (Alicante). 4Servicio de Cardiología, Hospital de Torrevieja (Alicante).
- 5016-10. UTILIDAD DEL ESTUDIO GENÉTICO EN LA MUERTE SÚBITA CON HALLAZGOS HISTOPATOLÓGICOS INESPECÍFICOS EN LA AUTOPSIA
- Mercedes Iglesias1, Tomás Ripoll Vera1, Jorge Álvarez Rubio1, Consuelo Pérez Luengo2, Damián Heine Suñer3, Bernardino Barceló Martín3, Susana Moyano Corvillo4, Concepción Dasi Martínez4, Juan Carlos Canós Villena4 y Juan Carlos Borondo Alcázar4
1Hospital Universitario Son Llàtzer, Palma de Mallorca (Illes Balears). 2Instituto de Medicina Legal de las Islas Baleares, Palma de Mallorca (Illes Balears). 3Hospital Son Espases, Palma de Mallorca (Illes Balears). 4Instituto Nacional de Toxicología, Barcelona.
- 5016-11. EVENTOS EMBÓLICOS EN PACIENTES CON AMILOIDOSIS CARDIACA POR TRANSTIRETINA CON FIBRILACIÓN AURICULAR
- Silvia Vilches Soria1, Esther González López1, Fernando Domínguez Rodríguez1, Luis Enrique Escobar López1, Adrián Rivas Pérez1, Aitor Hernández1, Stefano Perlini2, Claudio Rapezzi3, Mathew Maurer4, Julian Gillmore5 y Pablo García Pavía1
1Hospital Universitario Puerta de Hierro, Majadahonda (Madrid). 2Universidad de Pavía (Italia). 3Universidad de Bolonia (Italia). 4Hospital Universitario de Columbia, Nueva York. 5Centro Nacional de Amiloidosis, Londres (Reino Unido).
- 5016-12. ANÁLISIS DE LAS DIFERENCIAS CLÍNICAS Y DIAGNÓSTICAS ENTRE LOS PACIENTES CON PATOLOGÍA AÓRTICA FAMILIAR SINDRÓMICA Y NO SINDRÓMICA
- Martín Negreira Caamaño1, Jesús Piqueras Flores2, Jorge Martínez del Río1, Manuel Muñoz García1, Daniel Águila Gordo1, Cristina Mateo Gómez1, María Inmaculada Vivo Ortega2 y María Arántzazu González Marín3
1Servicio de Cardiología. 2Unidad de Cardiopatías Familiares, Servicio de Cardiología. 3Servicio de Pediatría, Hospital General Universitario de Ciudad Real.
- 5016-13. IMPLICACIÓN DE MUTACIONES EN AKAP9 Y KCNH2 EN LA EXPRESIÓN CLÍNICA DEL SÍNDROME DE QT LARGO TIPO1
- Eduardo Arana-Rueda1, Carmen María González de la Portilla Concha1, Juan Acosta Martínez1, Manuel Frutos López1, María Luisa Peña Peña2, Marta Navarro Arenas3, Alonso Pedrote Martínez4 y Antonio Castellano5
1Unidad de Arritmias, Servicio de Cardiología, Hospital Universitario Virgen del Rocío, Sevilla. 2Unidad de Cardiopatía familiares, Hospital Universitario Virgen del Rocío, Sevilla. 3Hospital Universitario Nuestra Señora de la Merced, Osuna (Sevilla). 4Unidad de Arritmias, Servicio de Cardiología, Hospital Universitario Virgen del Rocío, Sevilla. 5Instituto de Investigación Biomédica de Sevilla.
- 5016-14. APLICACIÓN DEL NUEVO SCORE DE RIESGO DE MUERTE SÚBITA DE SIEIRA EN UNA POBLACIÓN CON SÍNDROME DE BRUGADA
- Lidia María Carrillo Mora, María Jesús Fernández Gil, Francisco Javier Lacunza Ruiz, Pablo Peñafiel Verdú, Carmen Muñoz Esparza, Ignacio Gil Ortega, David López Cuenca, Eva Cabrera Romero, María Sabater Molina y Juan Ramón Gimeno Blanes
Hospital Clínico Universitario Virgen de la Arrixaca, El Palmar (Murcia).
- 5016-15. PERFIL DE EXPRESIÓN DE MICRORNAS ASOCIADOS AL FENOTIPO EN PACIENTES DE SÍNDROME DE BRUGADA Y EN PORTADORES DE MUTACIÓN EN EL GEN SCN5A
- María Cebro Márquez1, Ricardo Lage Fernández2, María Brión Martínez3, Eva Ramos Luis4, Juan Ramón Gimeno Blanes5, José Ramón González Juanatey6, Isabel Moscoso Galán2 y Moisés Rodríguez Mañero6
1Centro Singular de Investigación en Medicina Molecular y Enfermedades Crónicas (CIMUS), Santiago de Compostela (A Coruña). 2Centro Singular de Investigación en Medicina Molecular y Enfermedades Crónicas (CIMUS), Santiago de Compostela (A Coruña), CIBERCV. 3Fundación Pública Galega de Medicina Xenómica, Santiago de Compostela (A Coruña), CIBERCV. 4Fundación Pública Galega de Medicina Xenómica, Santiago de Compostela (A Coruña). 5Hospital Clínico Universitario Virgen de la Arrixaca, Murcia, CIBERCV. 6Complexo Hospitalario Universitario de Santiago de Compostela (A Coruña), CIBERCV.
- 5016-16. GEN FLNC EN LA MIOCARDIOPATÍA HIPERTRÓFICA: UN ESFUERZO PARA LOGRAR UNA CLASIFICACIÓN MÁS PRECISA DE LAS VARIANTES
- Elías Cuesta Llavona, Rebeca Lorca Gutiérrez, Beatriz Díaz Molina, José Luis Lambert Rodríguez, Sara Iglesias Álvarez, Belén Alonso González, José Julián Rodríguez Reguero, Eliecer Coto García y Juan Gómez de Oña
Hospital Universitario Central de Asturias, Oviedo (Asturias).
Más comunicaciones de los autores
-
Contreras Mármol, Helena
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- 6072-531 - IMPACTO DE LAS NUEVAS GUÍAS EN EL PORCENTAJE DE PACIENTES DENTRO DE OBJETIVOS DE LDL EN PACIENTES EN PREVENCIÓN SECUNDARIA DENTRO DE UNA UNIDAD DE REHABILITACIÓN CARDIACA
- 5016-2 - CRIBADO ACTIVO Y SECUENCIAL DE CASOS ÍNDICES DE HIPERCOLESTEROLEMIA FAMILIAR A PARTIR DE DATOS ANALÍTICOS CENTRALIZADOS
- 6071-521 - ¿ES LA VALORACIÓN DE LA CLASE FUNCIONAL SEGÚN LA NYHA COMPARABLE A LA VALORACIÓN OBJETIVA SEGÚN EL CONSUMO DE OXÍGENO PICO?
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
- 6004-10 - MEJORÍA DE LA DISFUNCIÓN VENTRICULAR IZQUIERDA POR ANTRACICLINAS TRAS OPTIMIZACIÓN DE TRATAMIENTO
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
-
de Cabo Porras, Carlos
- 6033-345 - TRATAMIENTO ANTIDIABÉTICO EN PREVENCIÓN SECUNDARIA EN PACIENTES CON CARDIOPATÍA ISQUÉMICA
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
- 6072-531 - IMPACTO DE LAS NUEVAS GUÍAS EN EL PORCENTAJE DE PACIENTES DENTRO DE OBJETIVOS DE LDL EN PACIENTES EN PREVENCIÓN SECUNDARIA DENTRO DE UNA UNIDAD DE REHABILITACIÓN CARDIACA
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- 6071-521 - ¿ES LA VALORACIÓN DE LA CLASE FUNCIONAL SEGÚN LA NYHA COMPARABLE A LA VALORACIÓN OBJETIVA SEGÚN EL CONSUMO DE OXÍGENO PICO?
- 6004-10 - MEJORÍA DE LA DISFUNCIÓN VENTRICULAR IZQUIERDA POR ANTRACICLINAS TRAS OPTIMIZACIÓN DE TRATAMIENTO
-
Gadella Fernández, Alejandro
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- 6033-345 - TRATAMIENTO ANTIDIABÉTICO EN PREVENCIÓN SECUNDARIA EN PACIENTES CON CARDIOPATÍA ISQUÉMICA
- 6049-401 - NUESTRO DÍA A DÍA EN LA ELEVACIÓN DE TROPONINA I MEDIANTE RESONANCIA CARDIACA
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
- 6072-531 - IMPACTO DE LAS NUEVAS GUÍAS EN EL PORCENTAJE DE PACIENTES DENTRO DE OBJETIVOS DE LDL EN PACIENTES EN PREVENCIÓN SECUNDARIA DENTRO DE UNA UNIDAD DE REHABILITACIÓN CARDIACA
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
- 6071-521 - ¿ES LA VALORACIÓN DE LA CLASE FUNCIONAL SEGÚN LA NYHA COMPARABLE A LA VALORACIÓN OBJETIVA SEGÚN EL CONSUMO DE OXÍGENO PICO?
- 6004-10 - MEJORÍA DE LA DISFUNCIÓN VENTRICULAR IZQUIERDA POR ANTRACICLINAS TRAS OPTIMIZACIÓN DE TRATAMIENTO
-
Rodríguez Padial, Luis
- 6005-66 - PERSPECTIVA DE MINOCA EN LA ACTUALIDAD, ¿CÓMO SEGUIMOS ACTUANDO?
- 6072-531 - IMPACTO DE LAS NUEVAS GUÍAS EN EL PORCENTAJE DE PACIENTES DENTRO DE OBJETIVOS DE LDL EN PACIENTES EN PREVENCIÓN SECUNDARIA DENTRO DE UNA UNIDAD DE REHABILITACIÓN CARDIACA
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
- 6004-10 - MEJORÍA DE LA DISFUNCIÓN VENTRICULAR IZQUIERDA POR ANTRACICLINAS TRAS OPTIMIZACIÓN DE TRATAMIENTO
- 6083-606 - ASOCIACIÓN DE LA ECTOPIA SUPRAVENTRICULAR DURANTE LOS PROCEDIMIENTOS DE CRIOABLACIÓN CON RECURRENCIAS DE FIBRILACIÓN AURICULAR
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
- 5020-6 - INCIDENCIA DE INSUFICIENCIA CARDIACA EN PACIENTES CON DIABETES MELLITUS TIPO 2. RESULTADOS A 1 AÑO DE SEGUIMIENTO EN EL ESTUDIO DIABET-IC
- 5016-2 - CRIBADO ACTIVO Y SECUENCIAL DE CASOS ÍNDICES DE HIPERCOLESTEROLEMIA FAMILIAR A PARTIR DE DATOS ANALÍTICOS CENTRALIZADOS
- 6066-499 - DETECCIÓN REMOTA PRECOZ DE DESCOMPENSACIÓN DE INSUFICIENCIA CARDIACA EN PORTADORES DE DAI-TRC
- 6009-113 - MINOCA: ¿EXISTEN DIFERENCIAS POR GÉNERO?
- 6071-521 - ¿ES LA VALORACIÓN DE LA CLASE FUNCIONAL SEGÚN LA NYHA COMPARABLE A LA VALORACIÓN OBJETIVA SEGÚN EL CONSUMO DE OXÍGENO PICO?
- 6033-345 - TRATAMIENTO ANTIDIABÉTICO EN PREVENCIÓN SECUNDARIA EN PACIENTES CON CARDIOPATÍA ISQUÉMICA
- 6079-591 - PAPEL DEL HOLTER INSERTABLE EN LA DETECCIÓN DE FIBRILACIÓN AURICULAR EN PACIENTES CON ICTUS CRIPTOGÉNICO: EXPERIENCIA EN UN CENTRO
- 6078-567 - ALERTAS EN EL ALGORITMO HEART LOGIC EN PACIENTES PORTADORES DE DAI-TRC EN SEGUIMIENTO REMOTO: CAUSAS Y DESENCADENANTES
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
-
Sabatel Pérez, Fernando
- 5016-2 - CRIBADO ACTIVO Y SECUENCIAL DE CASOS ÍNDICES DE HIPERCOLESTEROLEMIA FAMILIAR A PARTIR DE DATOS ANALÍTICOS CENTRALIZADOS
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
-
Sánchez-Prieto Castillo, Joaquín
- 5016-2 - CRIBADO ACTIVO Y SECUENCIAL DE CASOS ÍNDICES DE HIPERCOLESTEROLEMIA FAMILIAR A PARTIR DE DATOS ANALÍTICOS CENTRALIZADOS
- 6005-44 - PATRONES DE TRATAMIENTO HIPOLIPEMIANTE E INERCIA TERAPÉUTICA, TRAS OBJETIVAR ANALÍTICA ALTERADA, EN UNA POBLACIÓN CON HIPERCOLESTEROLEMIA GRAVE
- 5016-6 - ESTUDIO DE LAS ALTERACIONES GENÉTICAS EN PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR DEL ÁREA SANITARIA DE UN HOSPITAL DE TERCER NIVEL DETECTADOS MEDIANTE UNA NUEVA ESTRATEGIA DE CRIBADO SISTEMÁTICO PARTIENDO DE ANALÍTICA CENTRALIZADA PREEXISTENTE
- 6072-530 - CAMBIOS EN EL TRATAMIENTO Y GRADO DE CONTROL LIPÍDICO EN UNA COHORTE DE PACIENTES CON HIPERCOLESTEROLEMIA FAMILIAR
- 6004-10 - MEJORÍA DE LA DISFUNCIÓN VENTRICULAR IZQUIERDA POR ANTRACICLINAS TRAS OPTIMIZACIÓN DE TRATAMIENTO
- 6072-531 - IMPACTO DE LAS NUEVAS GUÍAS EN EL PORCENTAJE DE PACIENTES DENTRO DE OBJETIVOS DE LDL EN PACIENTES EN PREVENCIÓN SECUNDARIA DENTRO DE UNA UNIDAD DE REHABILITACIÓN CARDIACA
